我们发现每种测定法 100,000 细胞足以富集多种基因组修饰、转录因子和转录辅因子的靶基因座。然而,可以在不扩大珠、抗体或缓冲液的情况下将每个测定的细胞数量增加到 250,000。
我们已经证明,我们的 CUT&RUN Assay Kit #86652 仅适用于组蛋白修饰的 5,000-10,000 细胞和用于转录因子和辅因子的 10,000-20,000 细胞(参见图片)。
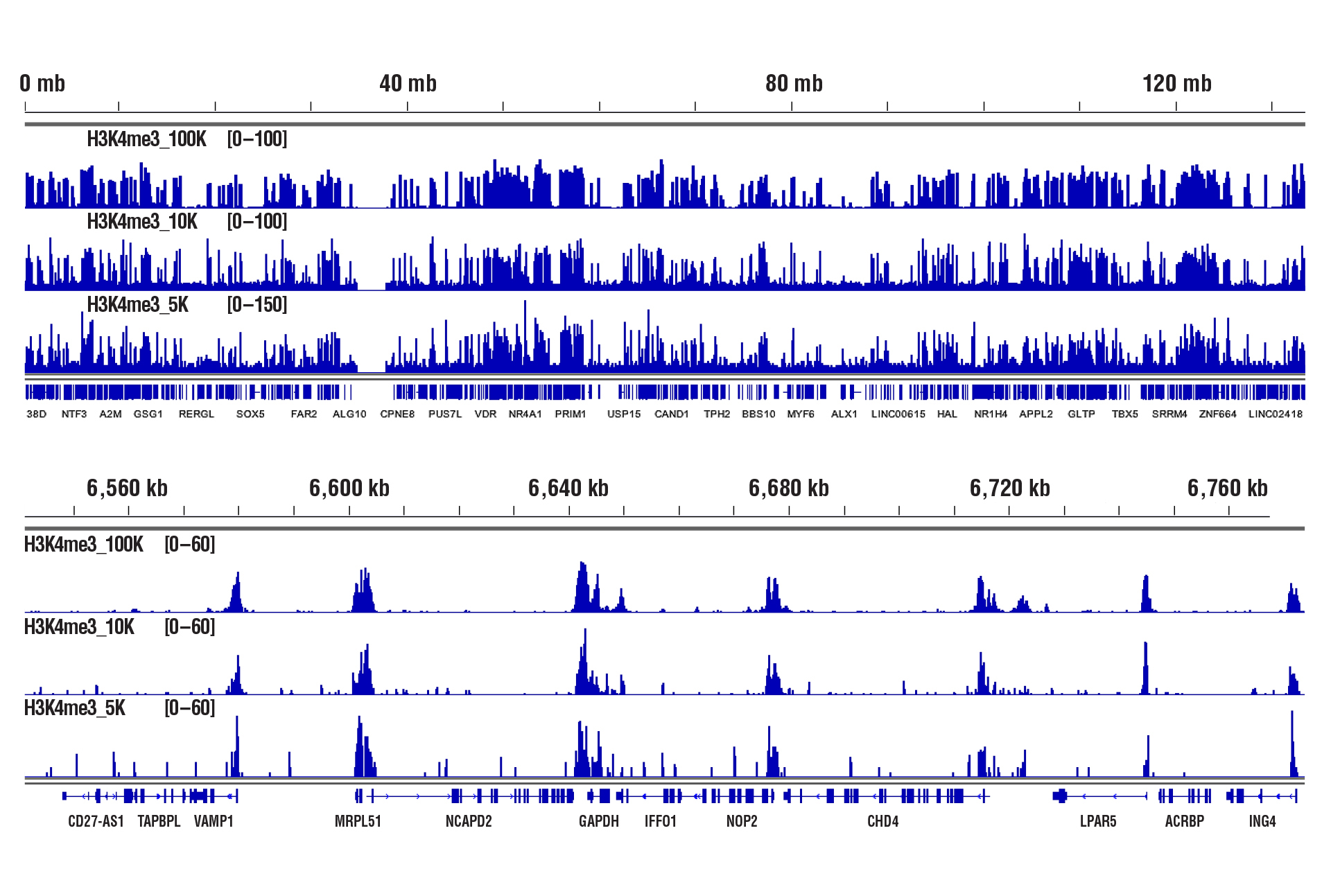
使用 CUT&RUN Assay Kit #86652 对 100,000、10,000或 5,000 HCT 116 细胞(按照指示)和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。上图显示跨染色体 12 的结合,而下图显示 GAPDH 基因(H3K4me3 的已知靶标)周围的富集。
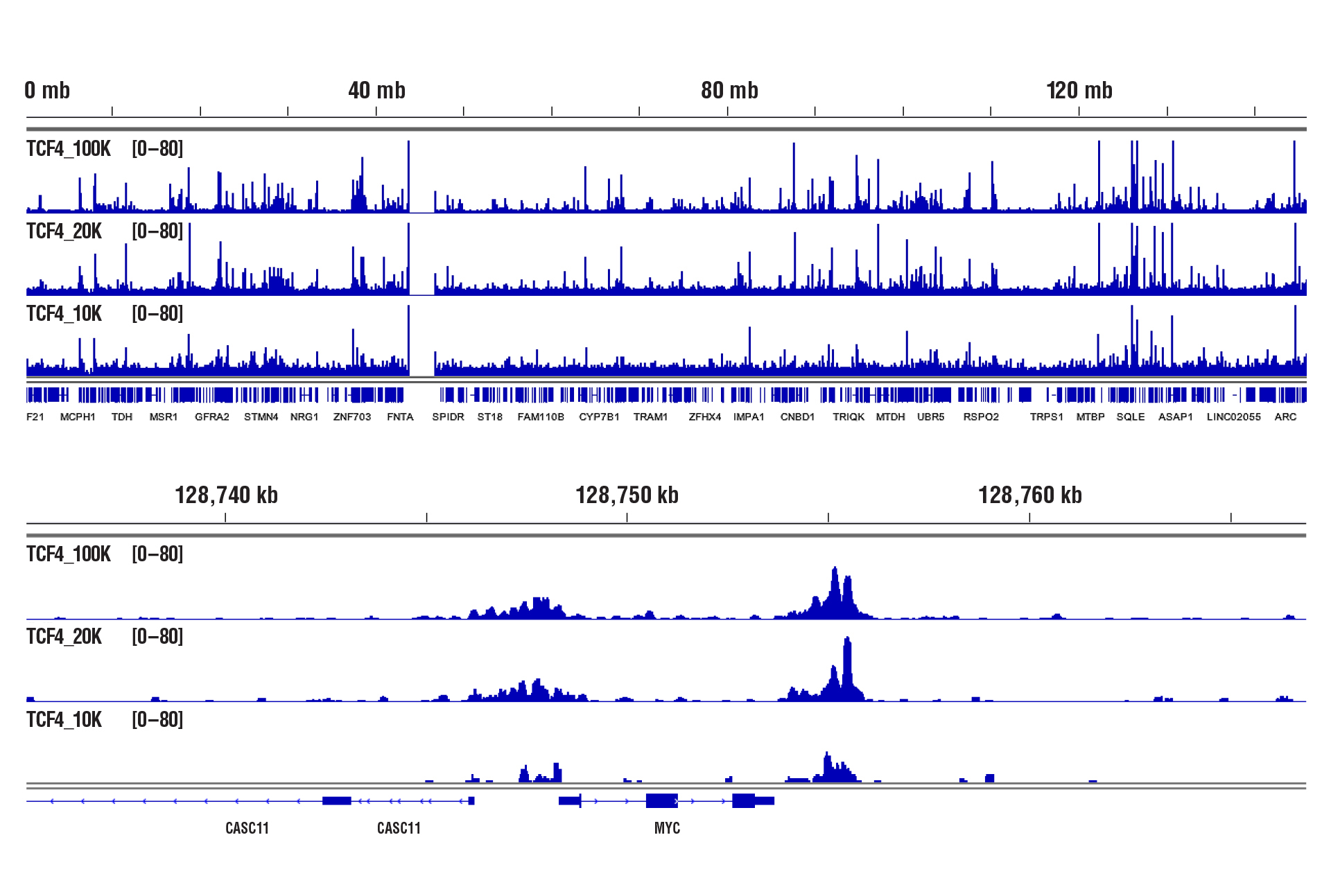
使用 CUT&RUN Assay Kit #86652 对 100,000、20,000或 10,000 HCT 116 细胞(按照指示)和 TCF4/TCF7L2 (C48H11) Rabbit mAb #2569 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。上图显示跨染色体 8 的结合,而下图显示围绕 MYC 基因(TCF4 的已知靶标)的富集。
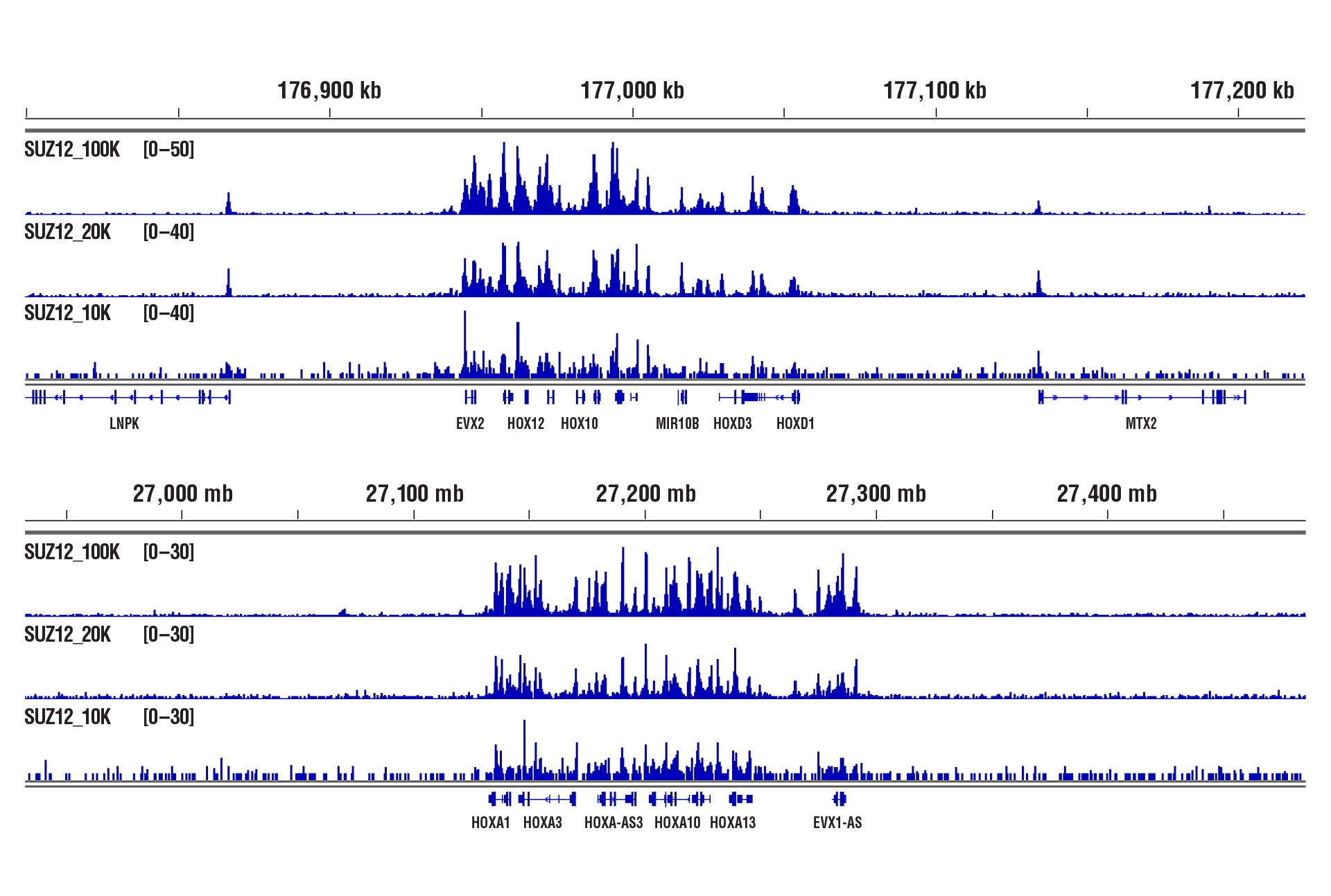
使用 CUT&RUN Assay Kit #86652 对 100,000、20,000或 10,000 NCCIT 细胞(按照指示)和 SUZ12 (D39F6) XP® Rabbit mAb #3737 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。该图显示了跨越 HoxD(上图)和 HoxA(下图)基因簇的结合,这些基因簇是 SUZ12 的已知靶标。
我们已对数十种培养的贴壁和悬浮细胞系(包括人和小鼠)进行了 CUT&RUN 分析。到目前为止,我们还没有注意到这些细胞系之间的性能有任何显著差异。
对于贴壁细胞系,首先需要将细胞从培养皿中分离。我们建议使用胰蛋白酶分离细胞。或者,您可以使用 Accutase,但是在离心过程中得到的细胞沉淀物不会很好地堆积,因此在洗涤步骤中您往往会丢失细胞。悬浮细胞要容易得多。只需使用血细胞计数器或自动细胞计数器对细胞进行计数,然后收集所需的细胞数量即可。我们没有观察到贴壁细胞和悬浮细胞的检测性能有显著差异。
可以。当细胞非常脆弱(在实验过程中很容易裂解)、细胞信号通路对用于细胞结合的伴刀豆球蛋白 A 珠敏感、目标蛋白与 DNA 结合较弱时,或者当您希望冷冻细胞沉淀物以备将来使用时,建议使用细胞固定。使用光固定实验步骤(0.1% 甲醛,2 分钟)对于使用固定细胞成功执行 CUT&RUN 分析至关重要。在测定结束时还需要反向交联步骤以从 DNA 中去除蛋白质。我们发现活性细胞在 CUT&RUN 测定中通常比固定细胞具有更好的性能,因此,我们建议尽可能使用活性细胞,除非在上述情况下需要固定(参见图片)
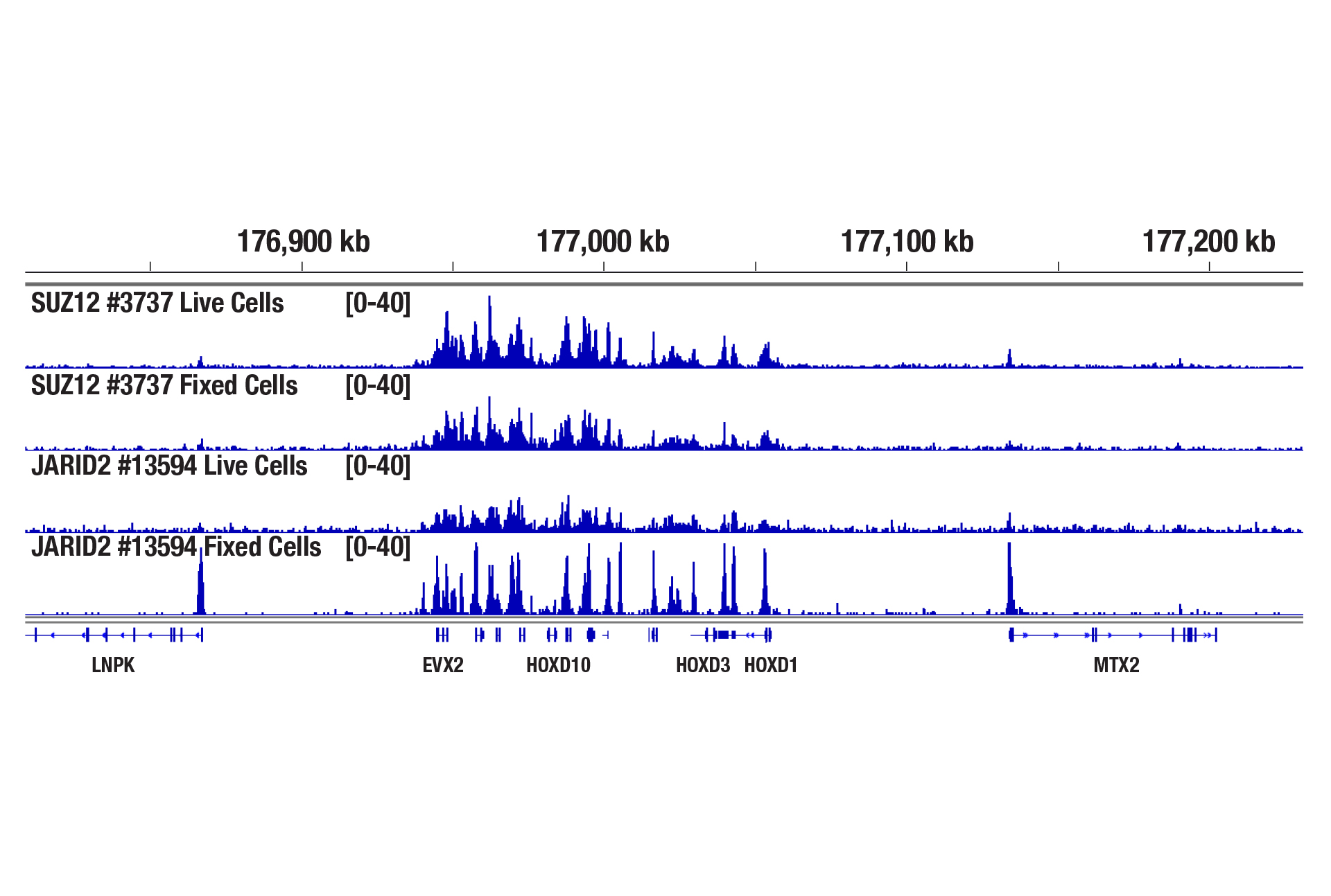
使用 CUT&RUN 检测试剂盒对 100,000 活性或固定的 NCCIT 细胞和 SUZ12 (D39F6) XP® Rabbit mAb #3737 或 JARID2 (D6M9X) Rabbit mAb #13594 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。该图显示了 SUZ12 和 JARID2 跨 HoxD 基因簇的结合。SUZ12(PRC2 复合物的核心成分)不需要固定,但会增加 JARID2(PRC2 复合物的附属成分)的富集。
可以。我们已经证明,我们的 CUT&RUN Assay Kit #86652 可用于 100,000 人 CD8+ 活 T 细胞,其中含有针对表观遗传调节因子和转录因子的抗体(参见图片)
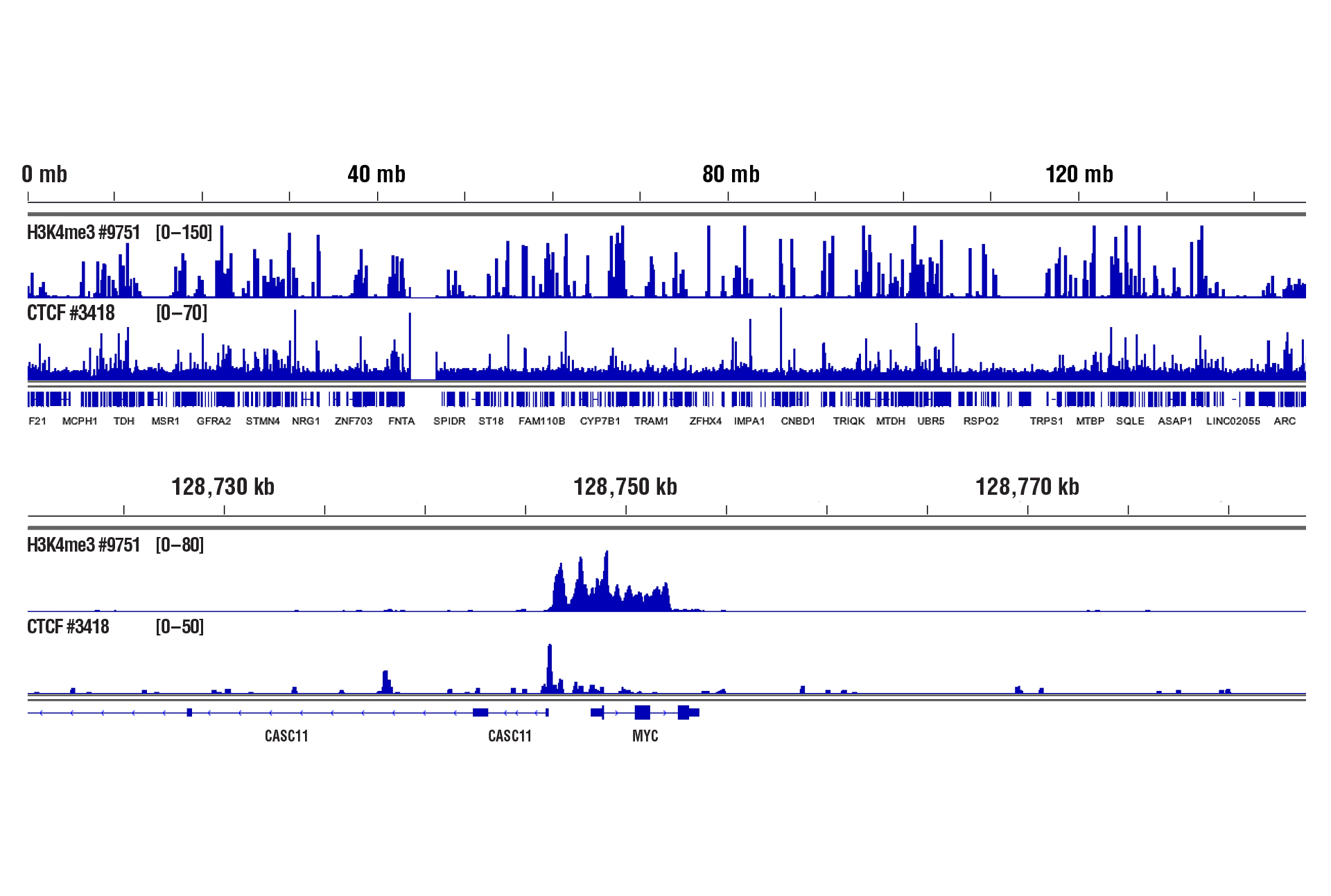
使用 CUT&RUN Assay Kit #86652 对 100,000 活的人 CD8+ T 细胞和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 或 CTCF (D31H2) XP® Rabbit mAb #3418 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。上图显示跨染色体 8 的结合,而下图显示 MYC 基因(H3K4me3 和 CTCF 的已知靶标)周围的富集。
可以。我们已经证明,我们的 CUT&RUN Assay Kit #86652 可用于小鼠肝脏、小鼠大脑和小鼠心脏组织,每次 CUT&RUN 反应只需 1 mg 组织。新鲜组织样本与组蛋白修饰和高丰度 DNA 结合因子(如 CTCF)一起使用。然而,为了用转录因子、辅因子和棘手组织类型获得最佳结果,应进行轻度至中等固定(0.1% 甲醛,2-10 钟)并且应使用略多点儿的起始材料 (2.5-5 mg) (参见附图)。固定的组织在用于 CUT&RUN 检测之前可以冷冻长达 6 个月。
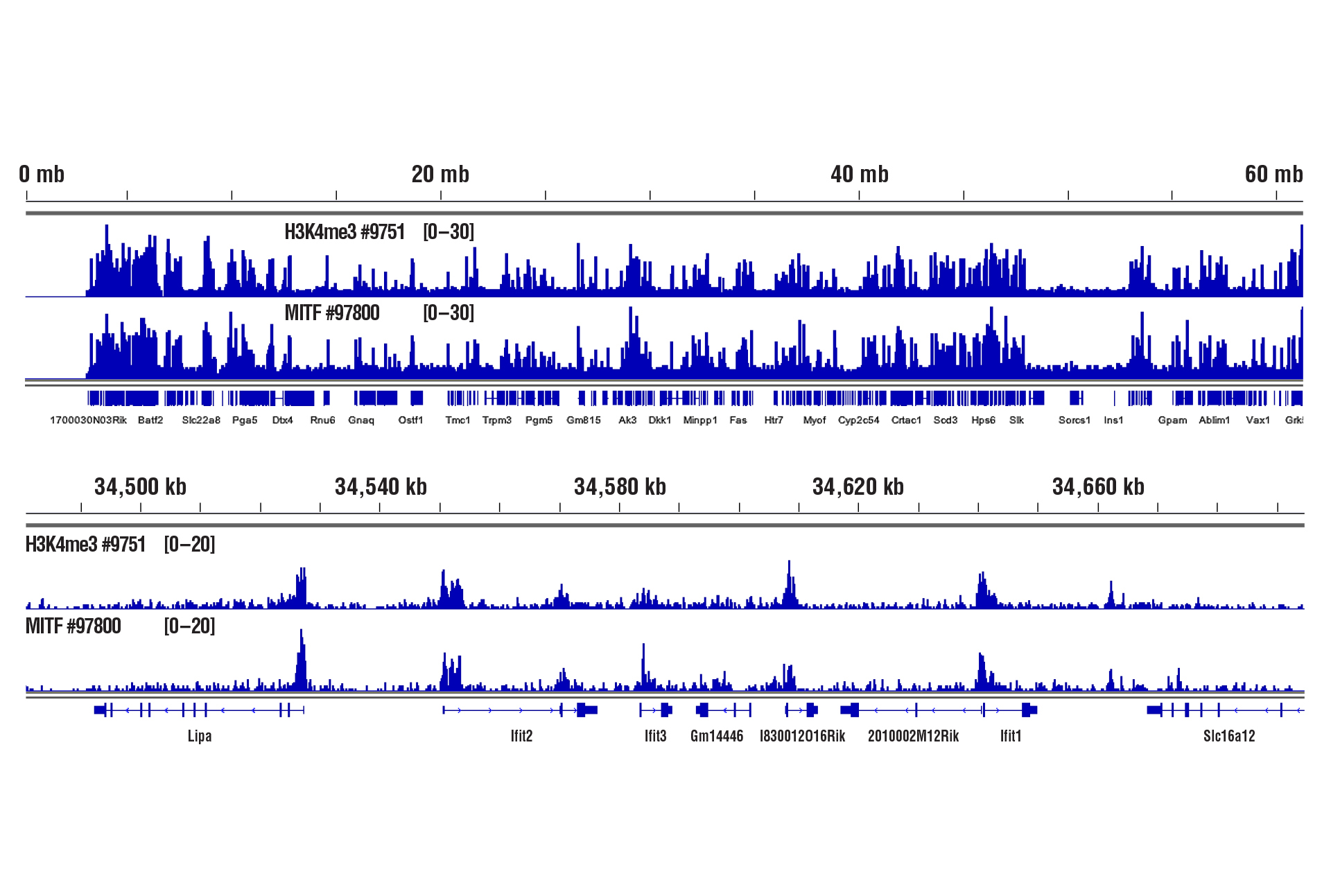
使用 CUT&RUN Assay Kit #86652 对 1 mg 轻微固定的小鼠心脏组织(0.1% 甲醛,2 分钟)、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 或 MITF (D3B4T) Rabbit mAb #97800 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。上图显示了 H3K4me3 和 MITF 跨染色体 19 的结合,而下图显示了 Ifit2 基因周围的结合。
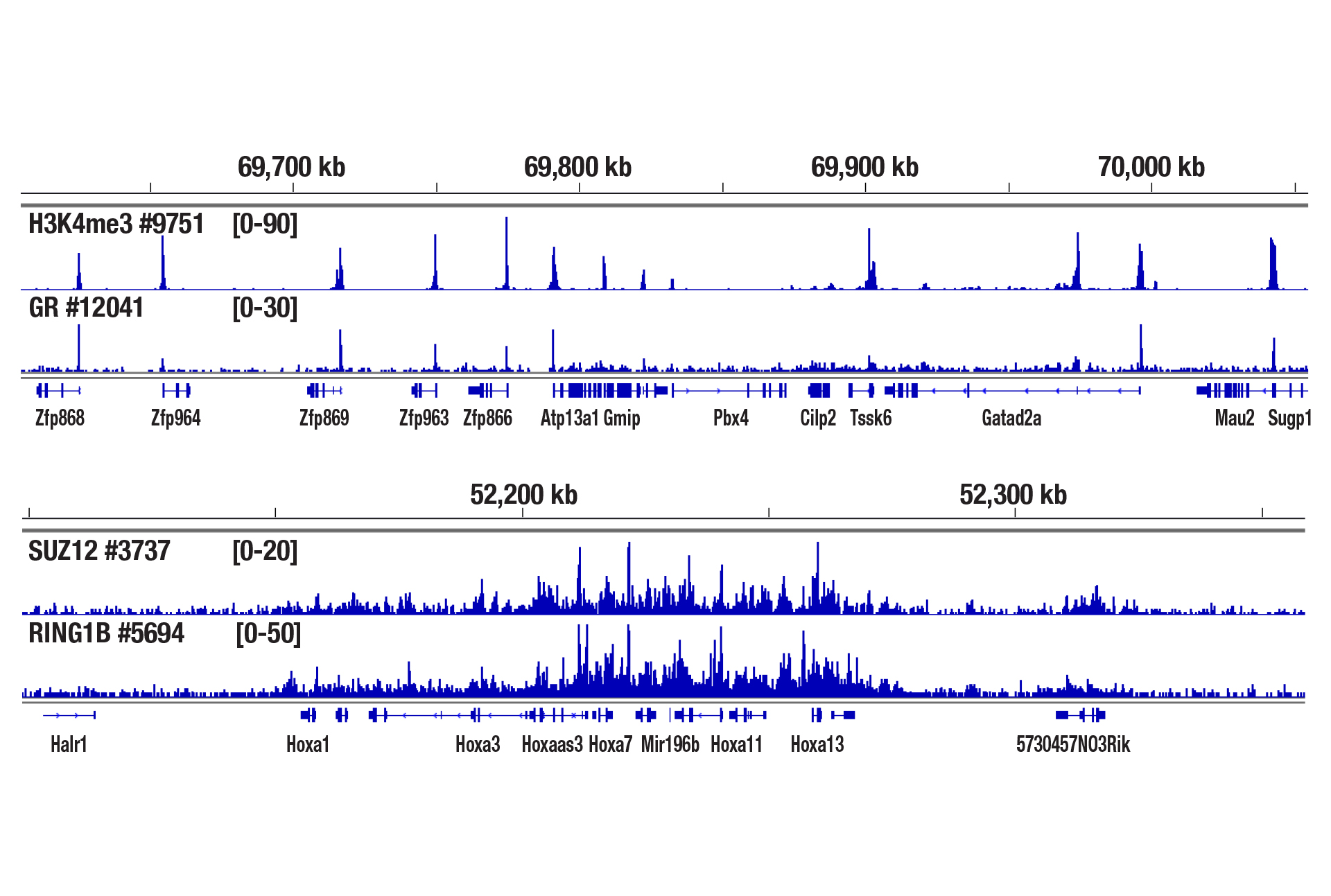
使用 CUT&RUN Assay Kit #86652 对 2.5 mg 中等固定的小鼠心脏组织(0.1% 甲醛,10 分钟)、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751、Glucocorticoid Receptor (D6H2L) XP® Rabbit mAb #12041、SUZ12 (D39F6) XP® Rabbit mAb #3737 或 RING1B (D22F2) XP® Rabbit mAb #5694 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。上图显示 H3K4me3 和糖皮质激素受体在 Zfp866 基因周围的结合,而下图显示 SUZ12 和 RING1B 跨 HoxA 基因簇的结合。
我们尚未使用植物细胞测试过 CUT&RUN 试剂盒。然而,Xiao-Yu Zheng 等人(2019;PMID30719569)描述了一种预处理植物组织并准备用于 CUT&RUN 的胞核的方法。我们的试剂盒可用于此胞核制备实验步骤的下游。在这种情况下,在 CUT&RUN 分析过程中可能不需要洋地黄皂苷进行细胞通透性,但是如果植物细胞不是 100% 同质性,它可能有助于进一步透化细胞膜。
Concanavalin A 磁珠可以很容易地凝集在一起,尤其是在细胞裂解时。确保您的细胞健康,并确保在细胞洗涤过程中非常轻柔地处理它们。我们还建议将细胞悬液与 Concanavalin A 磁珠的室温孵育时间限制为 5 分钟。在细胞珠结合、抗体结合和 pAG-MNase 酶结合的孵育期间替代转动或振摇试管,另一种选项是仅让试管在适当的孵育温度静置。这似乎有助于减少珠子结块且对最终结果无任何不利影响(参见下图)。尽管如此,我们的内部测试表明,珠子结块不会对 CUT&RUN 实验的最终结果产生负面影响。
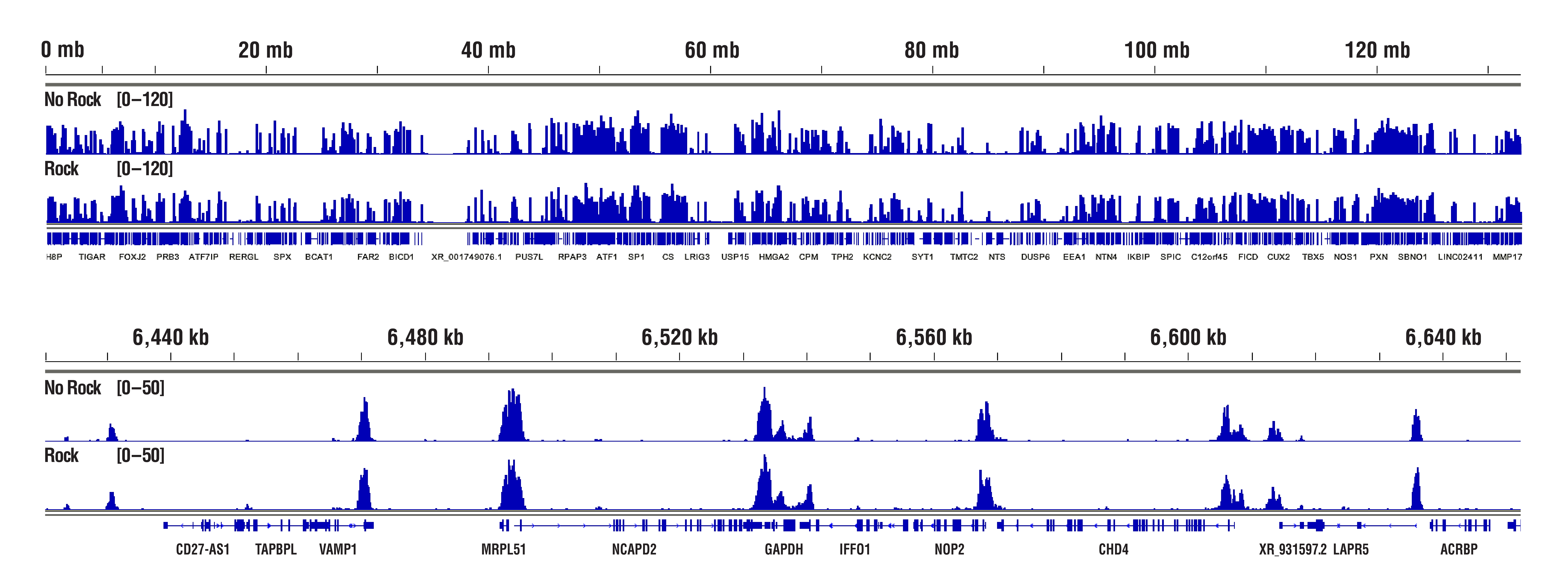
H3K4me3 数据突出显示“未振摇”样品对“振摇”样品的比较结果。 使用 CUT&RUN Assay Kit #86652 对 20,000 HCT 116 细胞和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 进行 CUT&RUN 检测。在细胞珠结合、抗体结合和 pAG-MNase 酶结合的孵育期间,将样品在适当的温度下静置(无振摇)或在旋转混匀仪(振摇)上振摇。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。各图显示跨染色体 12(上图)的结合,包括 H3K4me3 的已知靶基因 GAPDH(下图)。在无振摇实验步骤和振摇实验步骤之间未观察到信号强度或结合模式的显著差异。
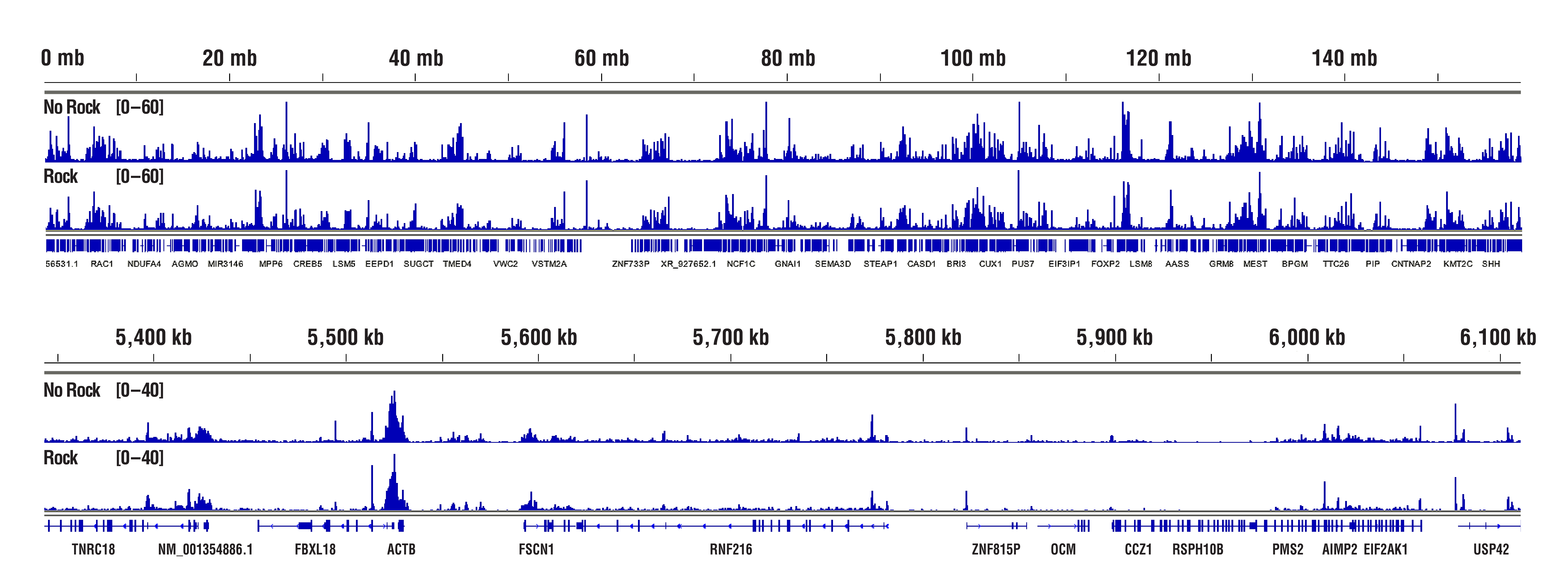
Rpb1 数据突出显示了“未振摇”样品对“已振摇”样品的比较结果。 使用 CUT&RUN Assay Kit #86652 对 100,000 HCT 116 细胞和 Phospho-Rpb1 CTD (Ser2) (E1Z3G) Rabbit mAb #13499 进行 CUT&RUN 检测。在细胞珠结合、抗体结合和 pAG-MNase 酶结合的孵育过程中,在适当的温度下,将样品静置(未振摇)或在旋转混匀仪(振摇)上进行摇晃。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。该图显示了染色体 7(上图)内的结合,包括 Phospho-Rpb1 的已知靶标基因 ACTB(下图)。在无振摇实验步骤和振摇实验步骤之间未观察到信号强度或结合模式的显著差异。
我们针对 CUT&RUN 分析而优化的消化条件可在多种细胞系(贴壁和悬浮液)上很好地发挥作用。另外,当增加或减少添加的 pAG-MNase 的量或消化时间时,我们也没有观察到染色质消化的变化。我们的观察结果与 Peter Skene 等人 (2018;PMID29651053)的出版物类似。也就是说,消化温度确实很重要。我们发现,与在 0°C 下消化相比,在 4°C 下消化可以显着增加信号强度,转录因子和辅因子的背景在可接受的范围内(参见图片)。
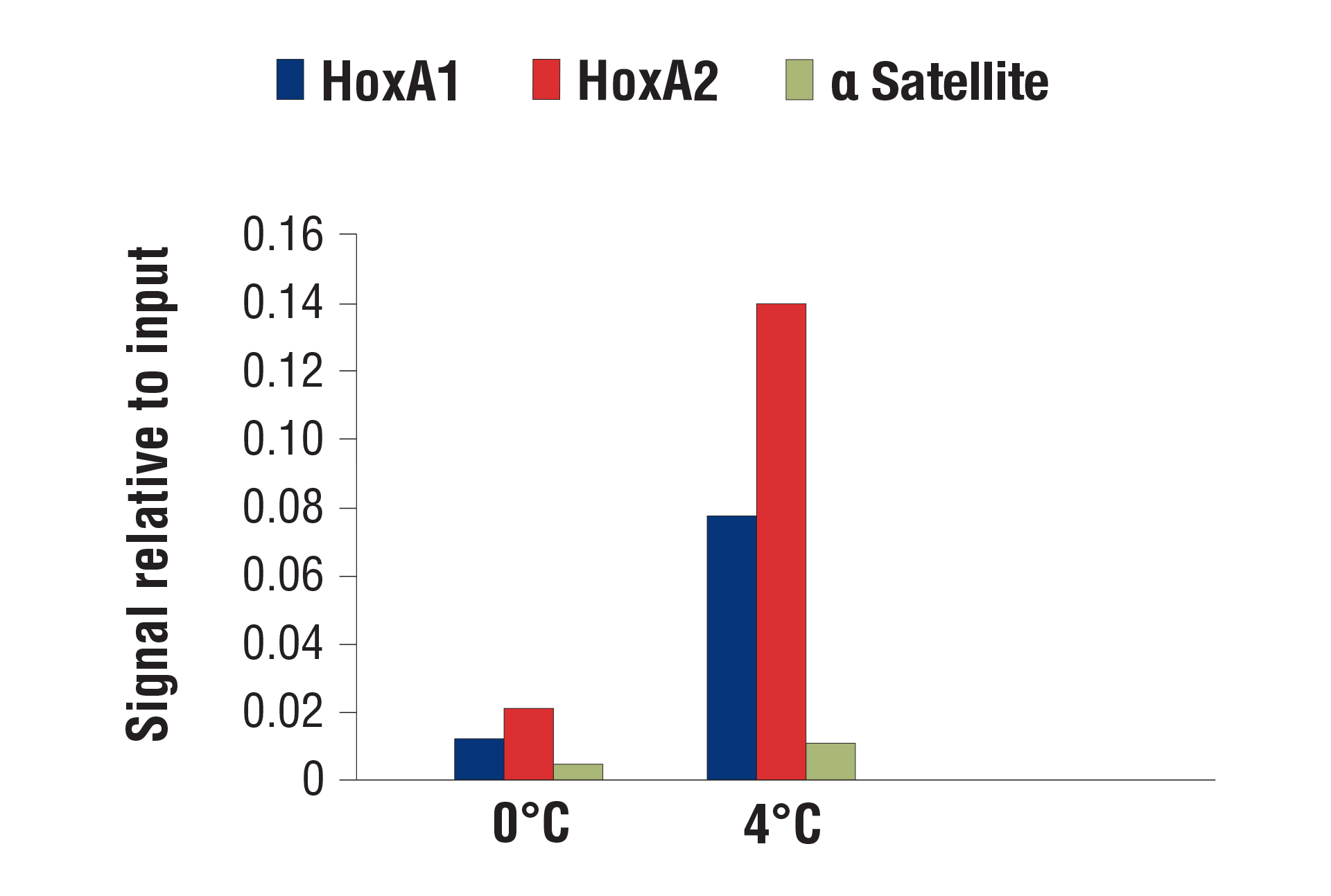
使用 CUT&RUN Assay Kit #86652 ,以 NCCIT 细胞和 SUZ12 (D39F6) XP® Rabbit mAb #3737 进行 CUT&RUN。如所示在 0°C 或 4°C 消化 DNA。使用 SimpleChIP® Human HoxA1 Intron 1 Primers #7707、SimpleChIP® Human HoxA2 Promoter Primers #5517 和 SimpleChIP® Human α Satellite Repeat Primers #4486 通过实时 PCR 对富集 DNA 进行定量分析。将每份样品中免疫沉淀的 DNA 的量表现为相对于所输入染色质总量(等于 1)的信号。
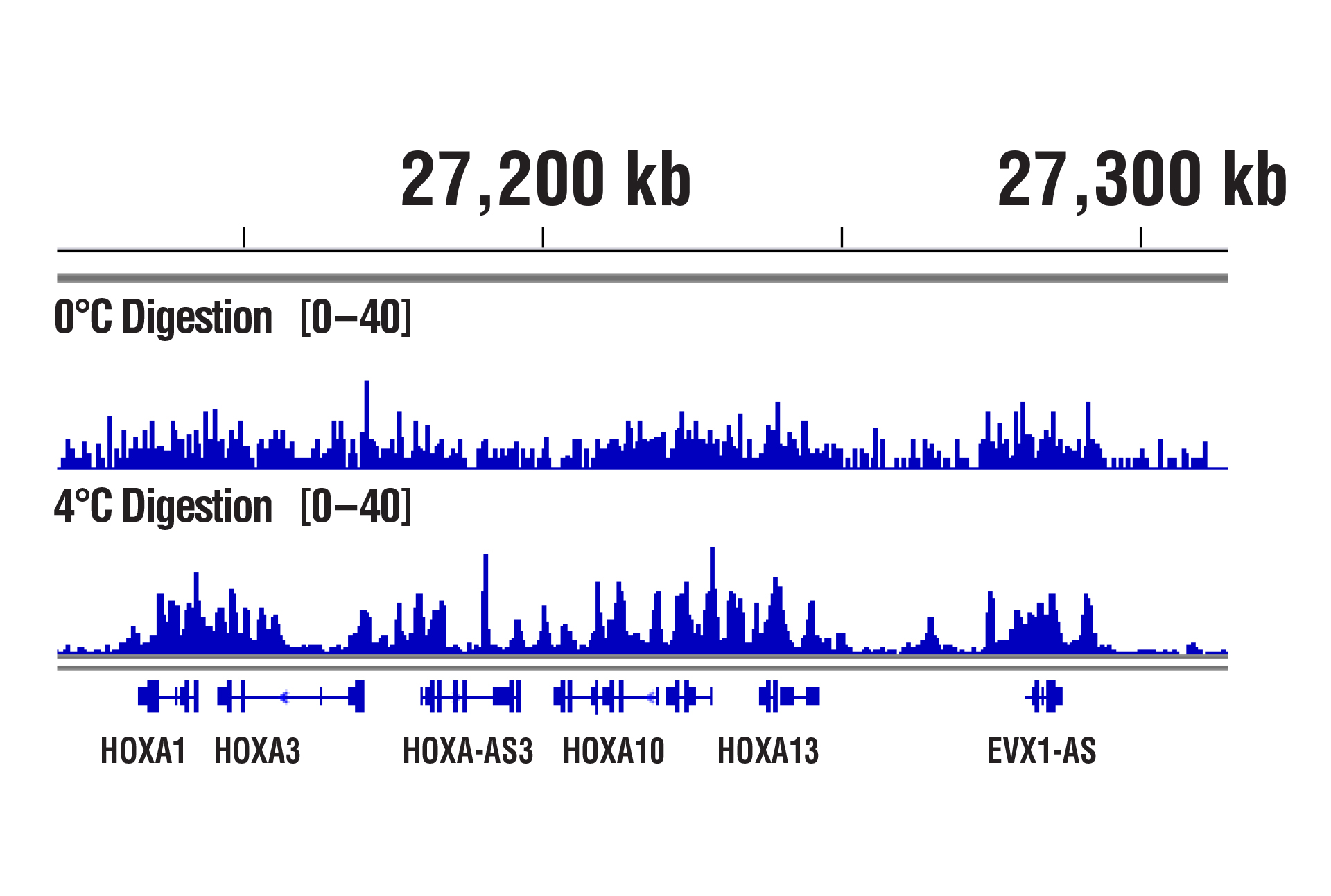
使用 CUT&RUN Assay Kit #86652 ,以 NCCIT 细胞和 SUZ12 (D39F6) XP® Rabbit mAb #3737 进行 CUT&RUN。如所示在 0°C 或 4°C 消化 DNA。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。该图显示跨各 HoxA 基因的结合。
是的,在整个实验过程中,所有缓冲液中都需要存在毛地黄皂苷,因为毛地黄皂苷引起的膜透化是可逆的。去除或降低缓冲液中毛地黄皂苷的量可能会干扰抗体和 pAG-MNase 进入细胞核。细胞对毛地黄皂苷浓度而不是毛地黄皂苷处理时间更敏感。如果您发现推荐的毛地黄皂苷量导致细胞大量裂解,您可以按照我们的 CUT&RUN 试剂盒 实验步骤中的附录 A 中所述优化检测中的毛地黄皂苷浓度 .
我们的 CUT&RUN 试剂盒经过优化,适用于整个细胞,并且适用于大量细胞系,包括贴壁和悬浮细胞。尽管通常不需要为 CUT&RUN 预分离细胞核,但我们的试剂盒应与预分离的胞核一起工作,因为伴刀豆球蛋白 A 珠与细胞和细胞核表面的糖蛋白结合。只有在洋地黄素不能有效渗透的细胞(如酵母细胞或植物细胞)含有细胞壁时,才需要预先分离细胞核。
否。在我们的测定中,我们没有观察到对常染色质或异染色质有任何偏见。在 CUT&RUN 中,靶标特异性抗体募集 pAG-MNase,并将消化引导至与该抗体直接相邻的染色质,无论是在常染色质中还是在异染色质中。在这种情况下,即使在较难接近的异染色质中,pAG-MNase 与染色质的有效束缚也允许发生消化。我们的 CUT&RUN 检测试剂盒 #86652已证明可与针对活性、可接近的常染色体组蛋白修饰(参见 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751)和激活转录因子(参见 NF-kB p65 [D14E12] XP® Rabbit mAb #8242))以及抑制性、难以接近的异色组蛋白修饰(参见 Tri-Methyl-Histone H3 [Lys27] [C36B11] #9733)以及与非活性异染色质相关的多梳阻遏物复合蛋白(参见 EZH2 [D2C9] XP® Rabbit mAb #5246 和 RING1B [D22F2] XP® Rabbit mAb #5694)的抗体一起使用(参见图片)。
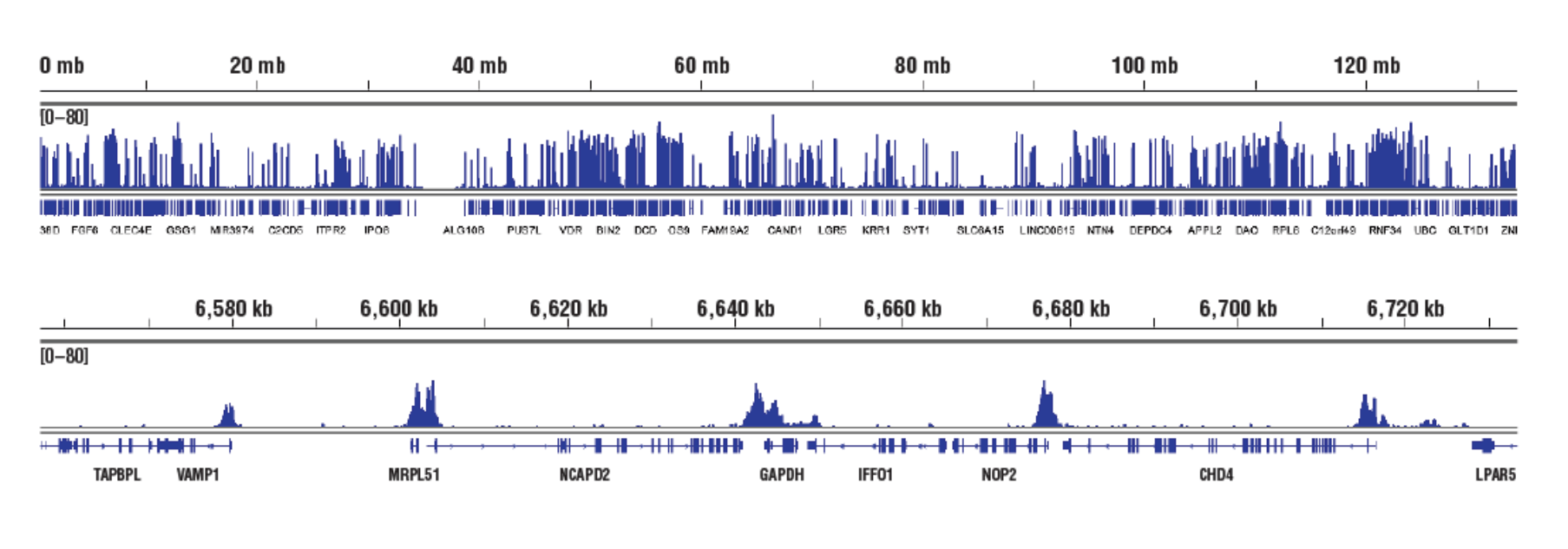
可接近的染色质,靶标:H3K4me3: 使用 CUT&RUN 检测试剂盒 #86652 对 HCT 116 细胞和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。各图显示跨染色体 12(上图)的结合,包括 H3K4me3 的已知靶基因 GAPDH(下图)。
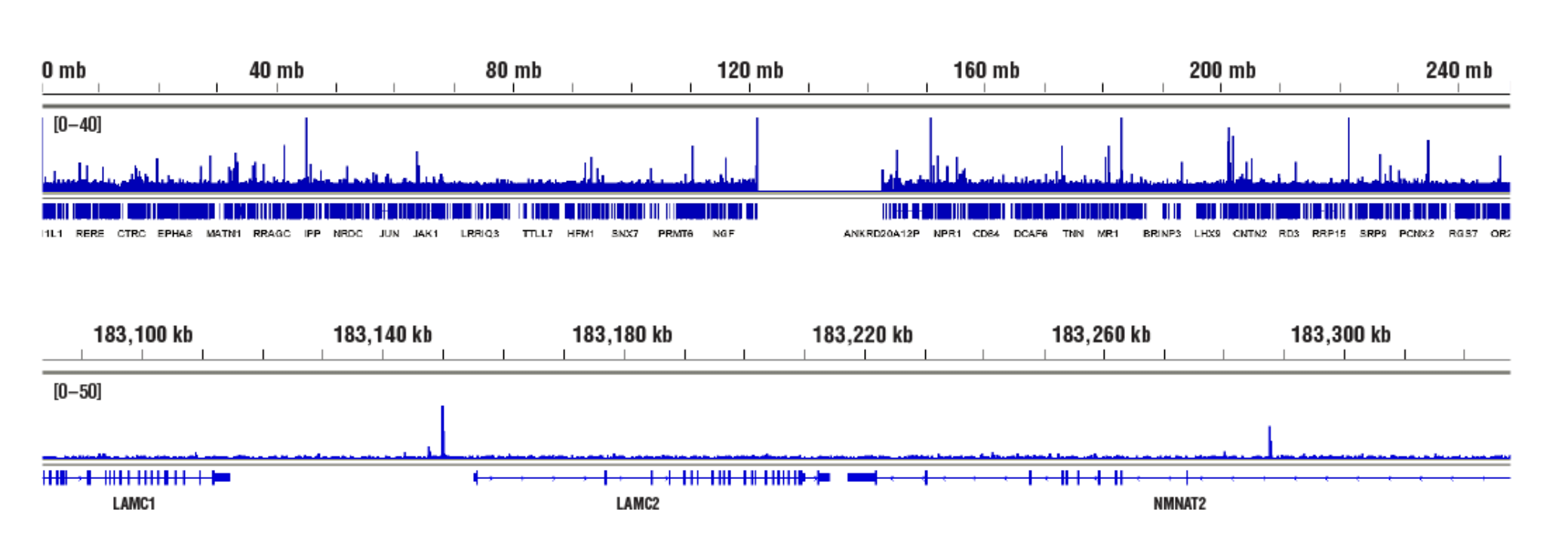
可接近的染色质,,靶标:NF-kB p65: 使用 CUT&RUN 检测试剂盒 #86652 对经 hTNF-α #8902(30 ng/ml,1 小时)和 NF-κB p65 (D14E12) XP® Rabbit mAb #8242 处理的 HeLa 细胞进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。结果图显示在染色体 1(上图)内的结合,包括 NF-κB p65 的已知靶基因 LAMC2(下图)。
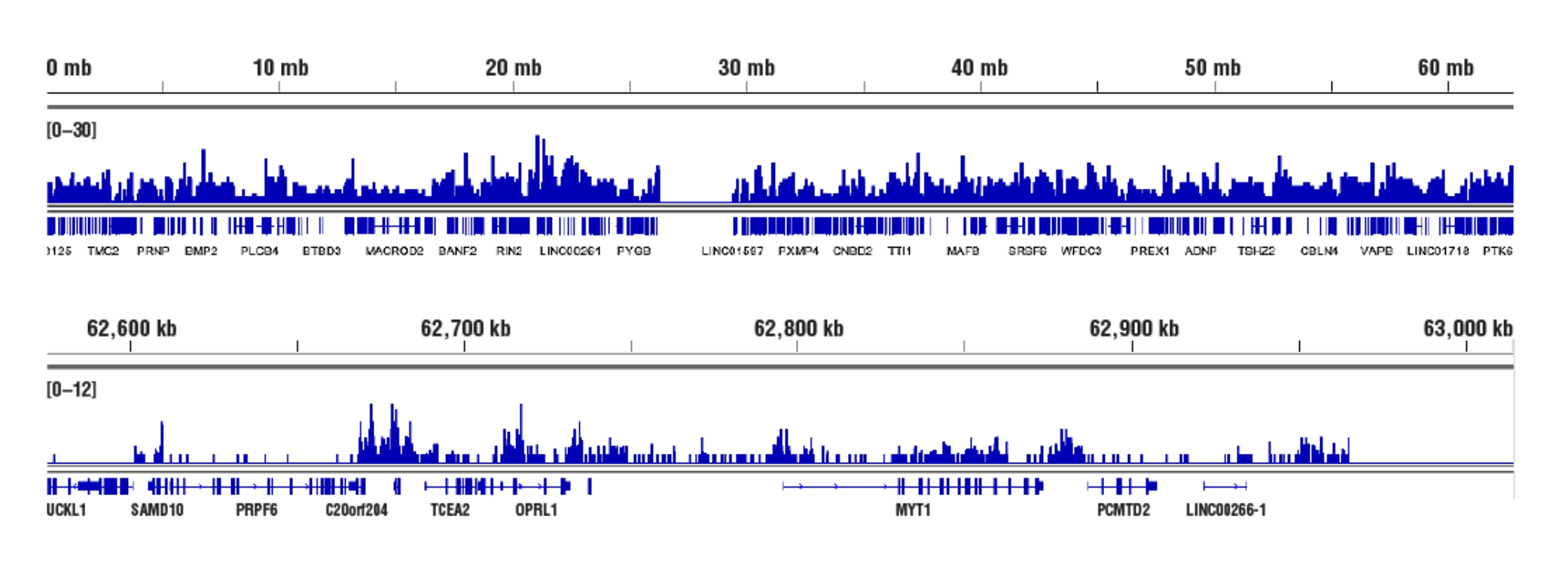
不可接近的异染色质,靶标:三甲基组蛋白 H3: 使用 CUT&RUN 检测试剂盒 # 86652,通过 HeLa 细胞和 Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb #9733 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。结果图显示在 20号染色体(上图)内的结合,包括 TCEA2 基因(下图)。
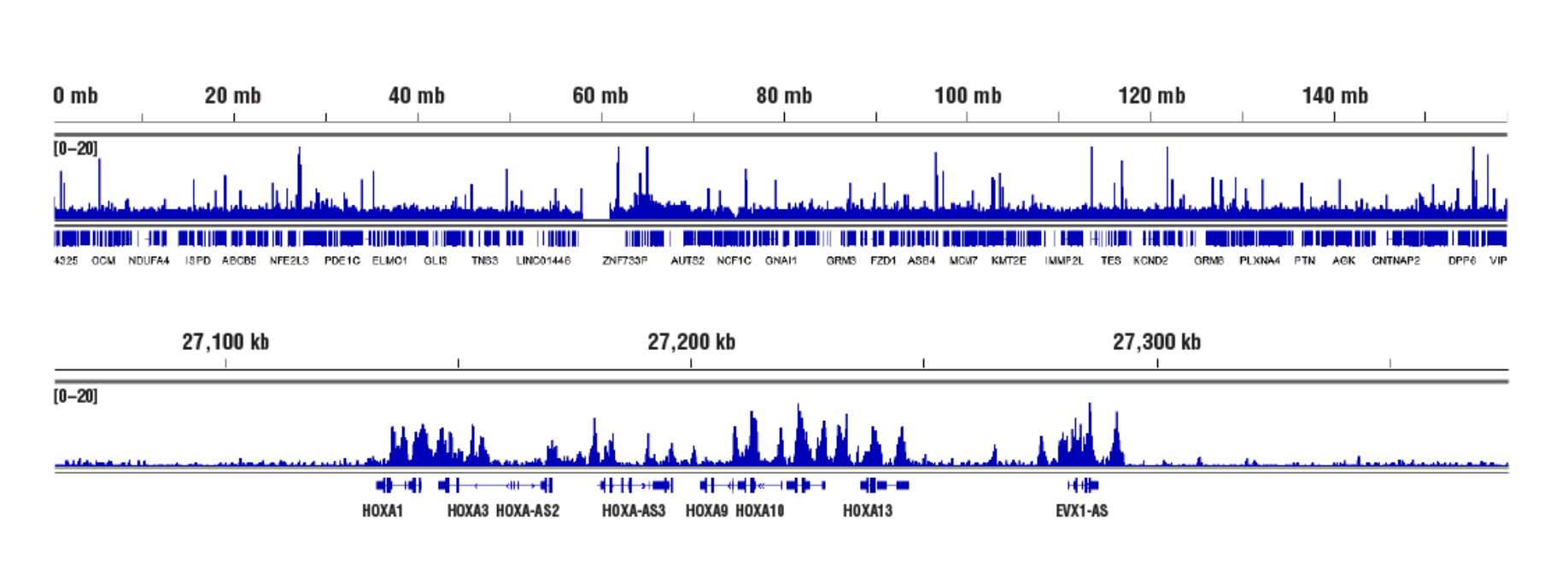
不可接近的异染色质,靶标:Ezh2: 使用 CUT&RUN 检测试剂盒 #86652,通过 NCCIT 细胞和 Ezh2 (D2C9) XP® Rabbit mAb #5246 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。结果图显示在染色体 7(上图)内的结合,包括 EZH2 靶标基因的已知基因簇 HoxA 基因(下图)。
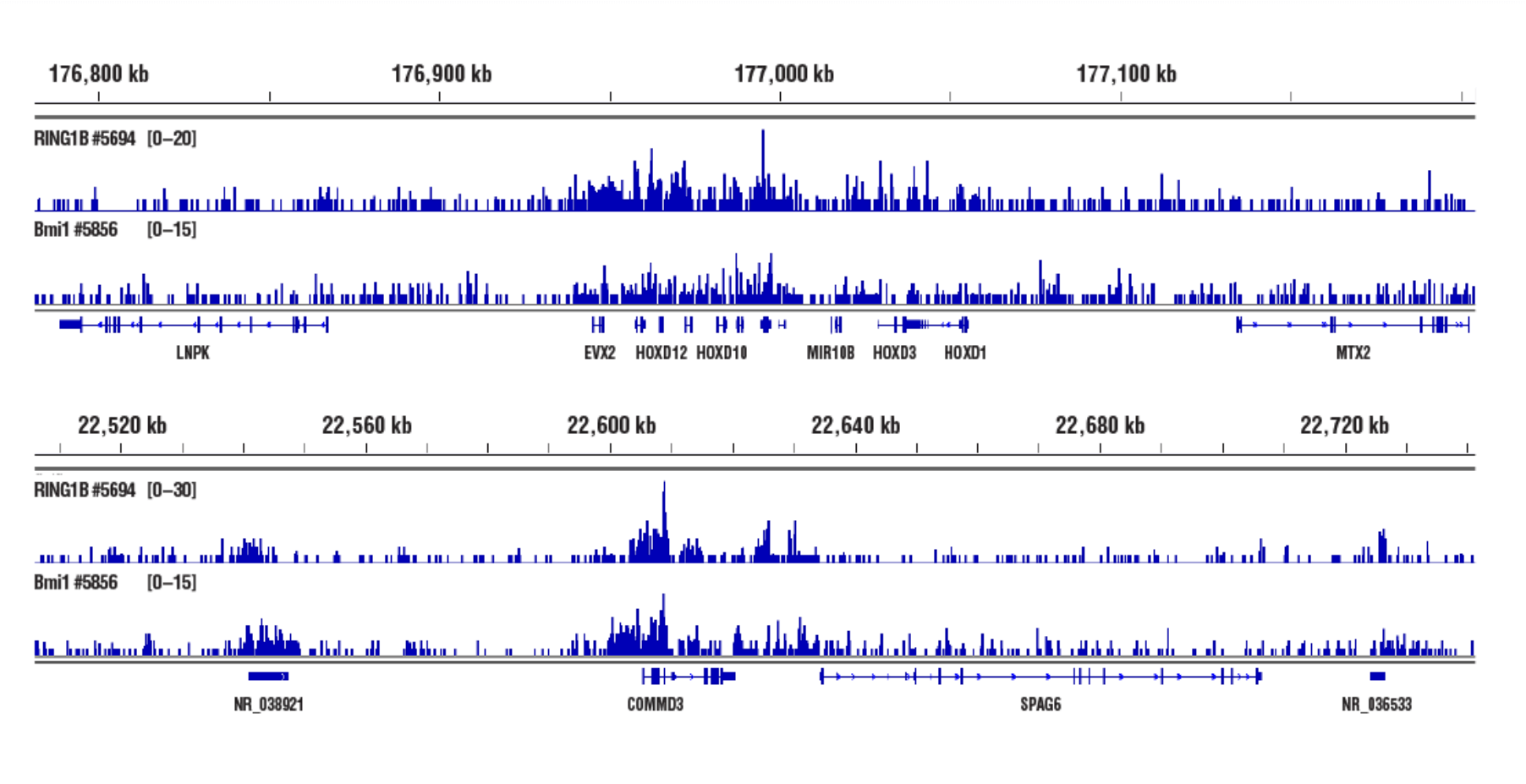
不可接近的异染色质,靶标:RING1B: 使用 CUT&RUN 检测试剂盒 #86652,通过 NCCIT 细胞和 RING1B (D22F2) XP® Rabbit mAb #5694 或 Bmi1 (D42B3) Rabbit mAb #5856 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。RING1B 和 Bmi1 均为 PRC1 组分。这些图显示在 HOXD(上图)和 COMMD3(下图)基因内结合。
无论是通过下游 qPCR 进行 CUT&RUN 分析还是进行 NG-seq 分析,我们始终建议使用经过 CUT&RUN 验证的阳性对照抗体,例如我们的 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 和我们的阴性对照抗体 Rabbit (DA1E) mAb IgG XP® Isotype Control Antibody #66362。这些抗体可以与我们的 SimpleChIP® Human RPL30 Exon 3 Primers #7014 或 SimpleChIP® Mouse RPL30 Intron 2 Primers #7015 配对,以表明您的 CUT&RUN 分析有效,无论所需的靶标特异性抗体的性能如何。所有这些对照物都包含在我们的 CUT&RUN Assay Kit #86652 中(参见图片)。
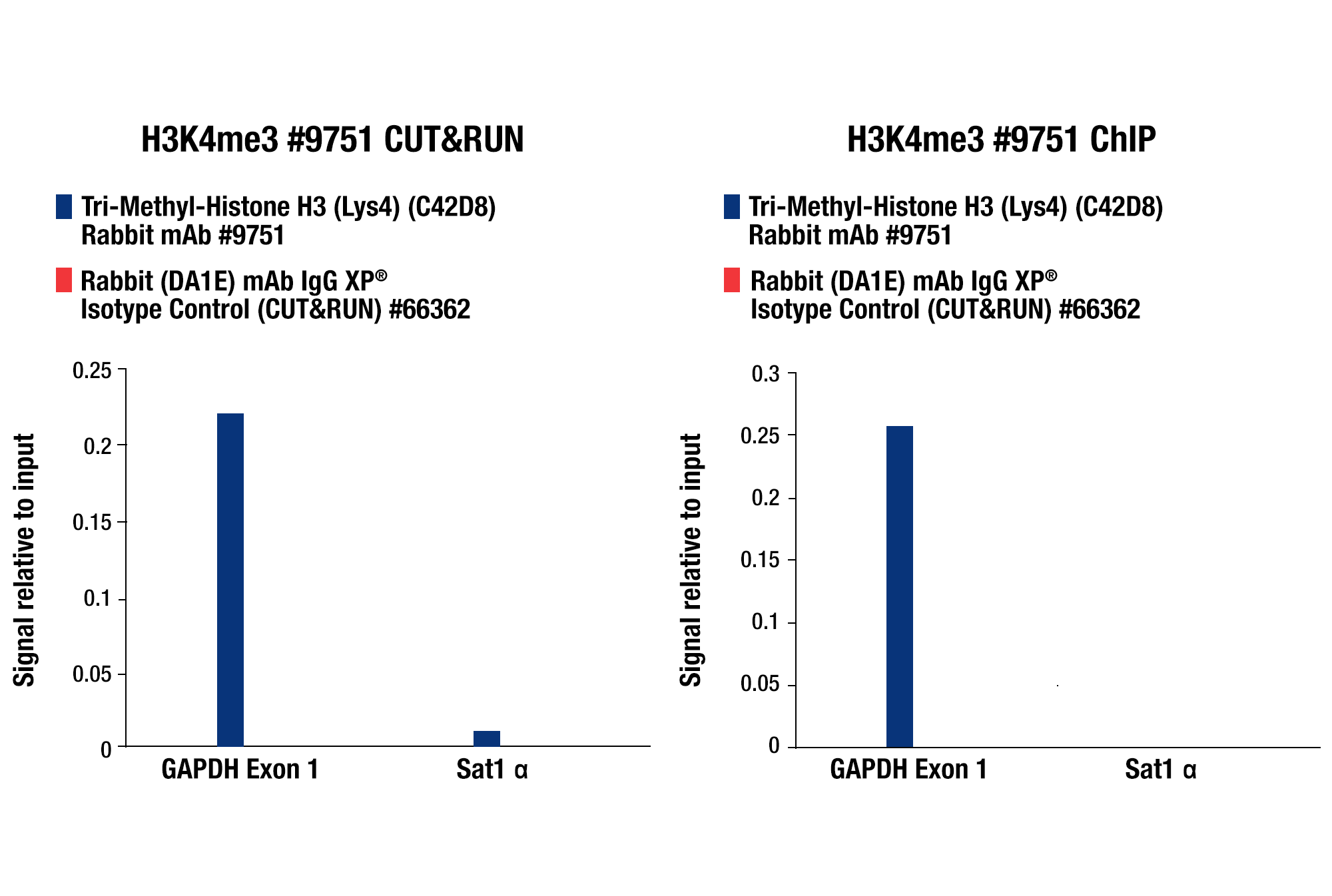
CUT&RUN qPCR 对照数据: 使用此 CUT&RUN 检测试剂盒 #86652(左小图)或 SimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005(右小图),对 HCT 116 细胞和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 或 Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362 进行 CUT&RUN 与 ChIP 测定。使用 SimpleChIP® Human GAPDH Exon 1 Primers #5516 和 SimpleChIP® Human α Satellite Repeat Primers #4486 进行实时 PCR 来对富集的 DNA 进行定量。将每份样品中免疫沉淀的 DNA 的量表现为相对于所输入染色质总量(等于 1)的信号。
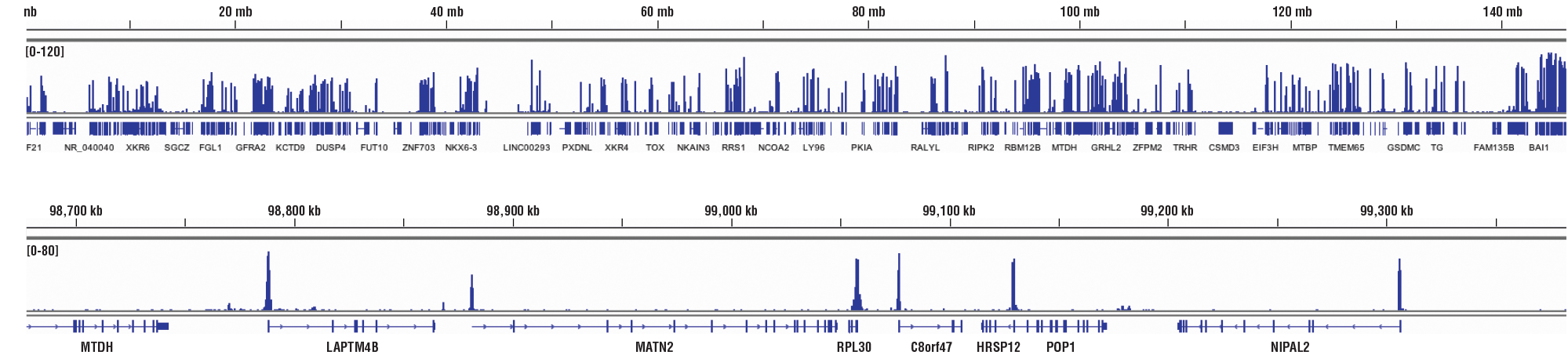
CUT&RUN NGS 对照数据: 使用此 CUT&RUN 检测试剂盒 #86652 或 SimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005,对 HCT 116 细胞和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 进行 CUT&RUN 和 ChIP 测定。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。上部小图比较了 H3K4me3 在染色体 12上的富集,而下部小图则比较了 H3K4me3 已知靶标 GAPDH 基因上的富集。input通道为 CUT&RUN 中的input样品。
我们建议从 ChIP 或 ChIP-seq 验证的抗体开始; 但是,我们发现并非所有经过 ChIP 验证的抗体都能在 CUT&RUN 分析中起作用。或者,Peter J. Skene 等人(2017;PMID28079019)建议,您可能想要尝试通过免疫荧光测定验证的抗体,因为 CUT&RUN 中的抗体结合发生在完整的胞核环境中,类似于免疫荧光测定中的抗体结合条件。
一些实验步骤利用 pAG-MNase 中污染大肠杆菌的 DNA 进行样品标准化。但是,这在切换 pAG-MNase 批次时会成为问题,因为每批酶都将具有不同量的污染 DNA。相反,我们建议使用单独的酵母刺突 DNA 样品。我们提供酵母刺突 DNA 样品和我们的 CUT&RUN 检测试剂盒 #86652 以及 CUT&RUN pAG-MNase 和刺突 DNA #40366, 每个批次都经过测试并针对 CUT&RUN 进行了优化。这种酵母刺突 DNA 为您的标准化工作提供了更好的批间一致性。
在消化后及 DNA 纯化之前,将指定量的预片段化酵母基因组 DNA 添加到每个 CUT&RUN 反应中。此刺突组件使您可以标准化样品之间的 DNA 纯化、qPCR、库制备和测序效率的差异。我们的 CUT&RUN 试剂盒中包含的实验步骤提供关于 qPCR 和 NG-seq 的刺突 DNA 对照的详细说明。重要的是要注意,qPCR 与 NG-seq 所需要的刺突 DNA 的量明显不同,因此请仔细阅读实验步骤。
当使用抗组蛋白修饰的抗体时,我们通常会看到片段大小小至 150 bp(单核小体大小),并且经常看到 DNA 片段大小的核小体阶梯(即150、300、450、600 bp)。当使用抗转录因子的抗体时,我们会看到一系列的 DNA 片段大小,大多数大于 35 bp(参见图片)。
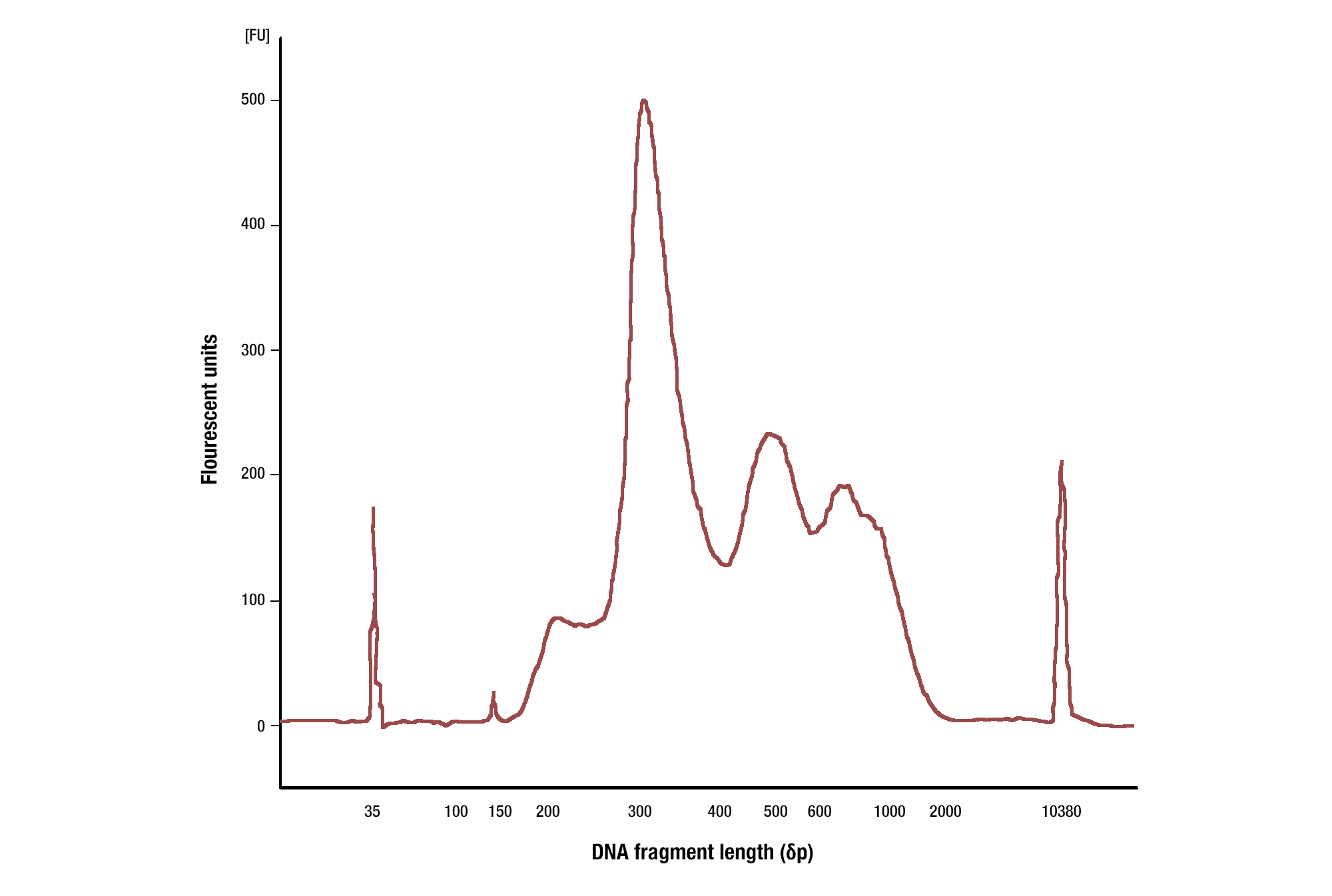
靶标:H3K4me3: 使用 HCT 116 细胞和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 进行 CUT&RUN 检测。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。使用 Agilent Bioanalyzer 分析文库中 DNA 片段的大小。构建期间向文库添加的接头和条码序列占了 140 bp 片段长度。如图所示,切除的 DNA 在单核小体上高度富集(峰值约为 300 bp)。
靶标:TCF4/TCF7L2: 使用 TCF4/TCF7L2 (C48H11) Rabbit mAb #2569 进行的 CUT&RUN 检测中使用苯酚/氯仿提取后再行乙醇沉淀方法来纯化 DNA。使用一台Bioanalyzer (Agilent Technologies) 分析文库中 DNA 片段的大小。构建期间向文库添加的接头和条码序列占了 140 bp 片段长度。
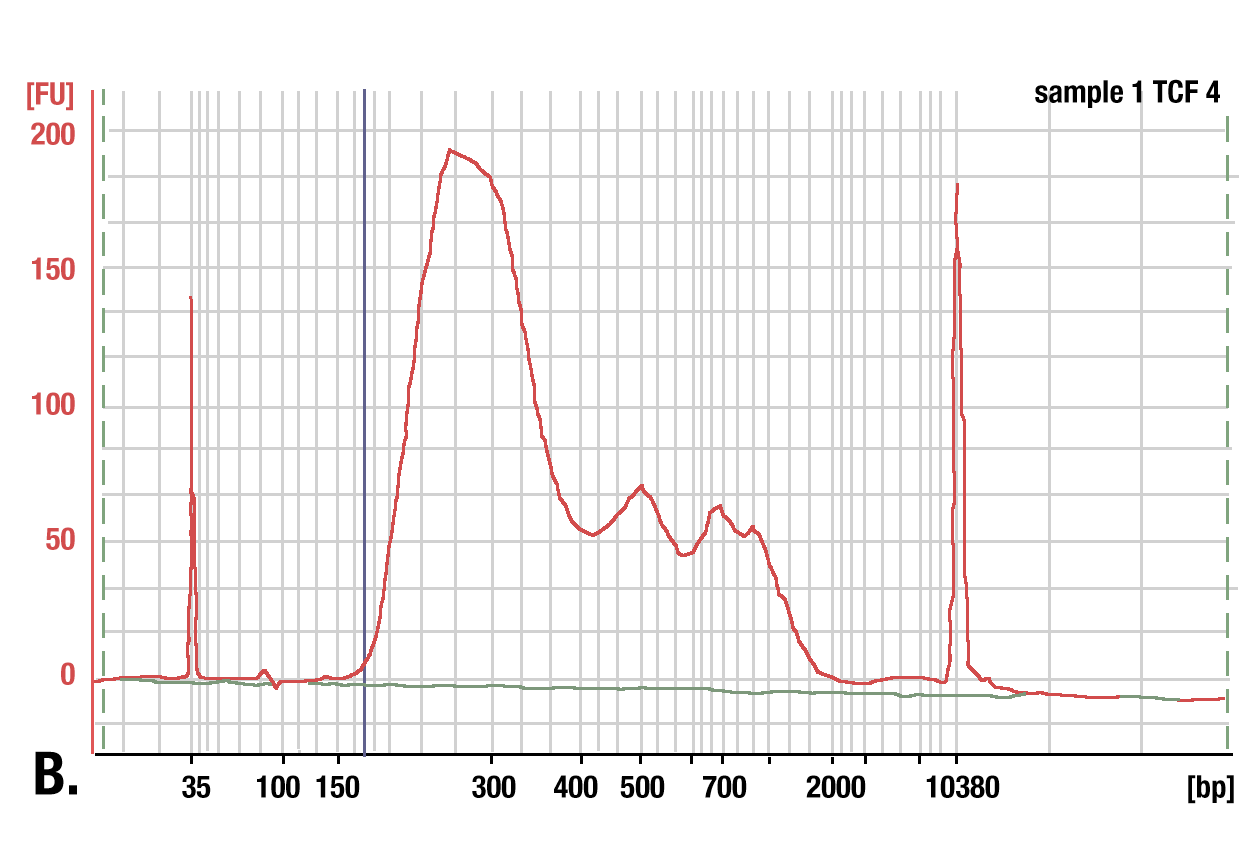
我们建议使用我公司 DNA 纯化缓冲液和离心柱( ChIP 和 CUT&RUN)#14209 纯化您的 CUT&RUN DNA。这些旋转柱为纯化 CUT&RUN DNA 提供了一种简单而经济的方法。在并行比较中,我们的 DNA 旋转柱可有效回收大于或等于 35 bp 的 DNA 片段。虽然先用苯酚-氯仿抽提再用乙醇沉淀确实能更有效地回收较小的 DNA 片段,但在分析由两个样品制成的 DNA 库时,我们发现 DNA 色谱柱捕获了约 98% 的全部 CUT&RUN DNA 片段。在苯酚-氯仿和乙醇沉淀样品中发现的DNA片段中约 2% 的长度小于 35 bp。因此,我们的 DNA 旋转柱为纯化 CUT&RUN DNA 片段提供了一种简单、经济且耐用的方法。请注意,并非所有的 DNA 旋转柱都能捕获相同范围的 DNA 片段大小,因此,如果您使用其他供应商的 DNA 旋转柱,则需要确定这些柱保留的最小 DNA 片段大小,因为更大的片段大小临界值会对您的结果产生负面影响(参见图片)。
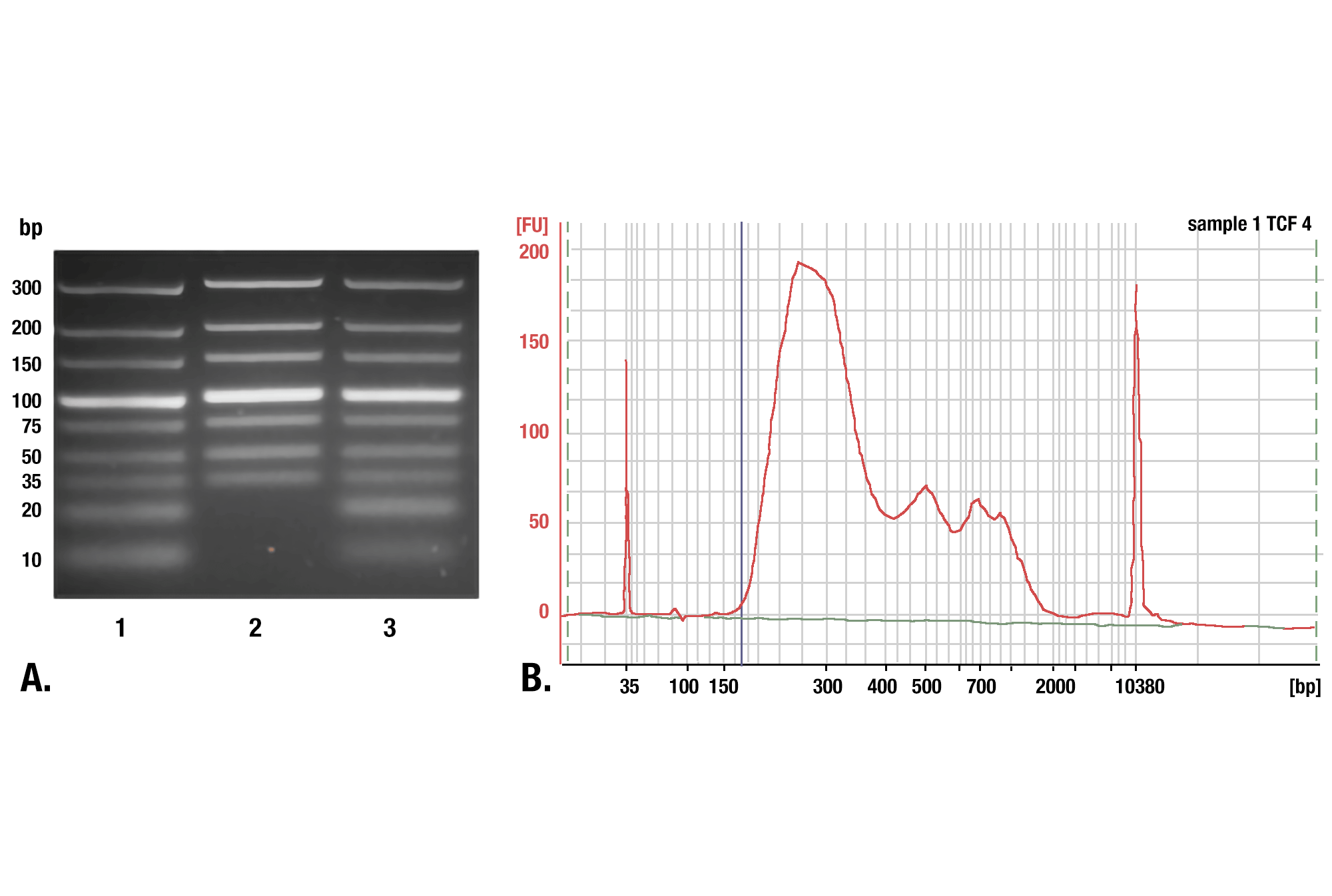
用于 DNA 纯化的苯酚/氯仿萃取与旋转柱: 比较使用离心柱或苯酚/氯仿提取后再行乙醇沉淀方法进行的 DNA 纯化。(A) 低量程 DNA 分子量标准(泳道 1,未纯化)使用 DNA Purification Buffers and Spin Columns (ChIP, CUT&RUN) #14209(泳道 2)进行纯化,或使用苯酚/氯仿提取后再行乙醇沉淀方法(泳道 3)进行纯化,并在 4% 琼脂糖凝胶电泳来进行分离。如图所示,苯酚/氯仿提取后再行乙醇沉淀能有效回收所有大小的 DNA 片段,而 DNA 离心柱仅能回收 ≥ 35 bp 的 DNA 片段。(B) 在使用 TCF4/TCF7L2 (C48H11) Rabbit mAb #2569 的 CUT&RUN 检测中使用苯酚/氯仿提取后再行乙醇沉淀方法来纯化 DNA。使用一台Bioanalyzer (Agilent Technologies) 分析文库中 DNA 片段的大小。构建期间向文库添加的接头和条码序列占了 140 bp 片段长度。因此,文库制备后,起始 35 bp 的 DNA 片段长度将变为 175 bp(图中用蓝色垂直线标注)。如所示, CUT&RUN 富集的总 DNA 片段中不到 2% 者短于 175 bp(起始长度 35 bp),这提示 DNA 纯化离心柱足以捕获 > 98% 的总 CUT&RUN DNA 片段。
我们的 CUT&RUN 试剂盒 与下游 qPCR 分析兼容。该试剂盒的实验步骤提供了生成输入 DNA 样本的说明和试剂,可用于通过 qPCR 定量您的 CUT&RUN DNA。对于 CUT&RUN DNA 的 qPCR 分析,我们建议设计引物,以产生长度为 60 至 80 bp 的扩增子。
您可以在 NG-seq 之前对 CUT&RUN 分析进行质量控制,方法是对您的富集 DNA 进行 qPCR 分析。我们的 CUT&RUN 试剂盒 与下游 qPCR 分析兼容。
使用下游 qPCR 分析进行 CUT&RUN 检测时,可以使用富含 IgG 抗体的正常样品作为阴性对照,并将靶标特异性抗体富集度计算为 IgG 背景的倍数。但是,IgG 样品通常包含极少量的 DNA,并且不能很好地指示您的靶标特异性抗体在测定中的作用。因此,我们建议生成并使用输入染色质 DNA 样品。如何生成输入染色质 DNA 样本在我公司 CUT&RUN 试剂盒实验步骤的第 V 部分作了描述。这样,您可以将目标特异性抗体 DNA 富集表达为总输入 DNA 的百分比,并直接确定靶标特异性抗体在测定中的作用情况。
Bioanalyzer 要求 5 到 10 ng DNA 以最佳分析 DNA 片段大小。然而,成功的 CUT&RUN 实验通常可以从 100,000 起始细胞中产生少于 1 ng 的 DNA。这种低产量在转录因子和辅因子分析中最为常见。我们发现这种低产量是可以的,并且仍然可以提供可靠的测序数据。在这些情况下,我们建议您继续进行 DNA 测序文库制备,然后使用生物分析仪分析您的 DNA 文库(DNA 文库的典型浓度为 10 至 40 ng/μl)。与其依赖 DNA 产量作为 CUT&RUN 实验的 QC 测量,我们建议在执行 NG-seq 之前通过 qPCR 分析您的 CUT&RUN 实验以查看一些已知目标基因的富集情况。
这取决于 DNA 库制备试剂盒的敏感性。每个反应的 CUT&RUN DNA 的典型产量为 0.6 至 6 ng。我公司 SimpleChIP56795® ChIP-seq DNA Library Prep Kit for Illumina® # 可用于低至 ng 的起始 DNA。如果起始量太低,您也可以将 PCR 循环数增加到 20,以增加库的扩增。
我们建议使用我公司 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 以及 CUT&RUN 或 ChIP DNA 产生 DNA 文库。该试剂盒应该配合我公司 Multiplex Oligos for Illumina (Dual Index Primers) (ChIP-seq, CUT&RUN) #47538 或 Multiplex Oligos for Illumina (Single Index Primers) (ChIP-seq, CUT&RUN) #29580 使用。
不,我们不建议在准备用于 CUT&RUN 或 ChIP-seq 的库时执行大小选择。根据我们的经验和客户反馈,大小选择可以显著降低 DNA 文库产率和多样性。如果您对大于 1kb 的大 DNA 片段有顾虑,则在文库制备期间扩增接头连接的 DNA 样品时,单纯减少延伸时间即可。我们发现将延伸时间减少到 10 至 15 秒大大减少了大于 1kb 的大片段的扩增,同时足以在 CUT&RUN 库制备过程中扩增较小和更理想的 DNA 片段。
在进行下游 NG-seq 分析的 CUT&RUN 检测时,可以使用富含 IgG 抗体的正常样品作为阴性对照。但是,我们从其他科学家那里听说过,正常的 IgG 抗体会产生非常低的多样性 DNA 库,并且在 CUT&RUN 检测中可能会产生看起来特别是基因组区域富集的现象,从而使 NG-seq 数据分析变得复杂。相反,我们建议生成并使用如 CUT&RUN 试剂盒的实验步骤第 V 部分 中所述的输入染色质 DNA 样品。此输入的 DNA 样本生成了一个更多样化的 DNA 库,并且可以更好地用作 NG-seq 分析的阴性对照。
如果没有超声仪并且需要进行下游 NG-seq 分析,请使用我公司实验步骤页面上所载的 微球菌核酸酶消化 CUT&RUN 输入 DNA 实验步骤。如果没有超声波仪并且需要进行 qPCR 分析,则未片段化的输入 DNA 可用于 qPCR 分析;然而,未片段化的输入 DNA 应使用苯酚/氯仿提取和乙醇沉淀进行纯化,因为未片段化的输入 DNA 的大小太大而无法使用 DNA 离心柱进行纯化。或者,酶切的输入 DNA 也可用于 qPCR 分析。
这一决定基于我们对数百份 CUT&RUN 样品的分析。我们发现低于 300 万的测序深度产生的峰太少,并且随着深度测序的增加,峰数增加。如果需要,可以将测序深度增加到每个样品 1000 万个读数。如果每份样品的测序深度大于 1500 万,则测序读数的重复率显著增加(参见图片)。
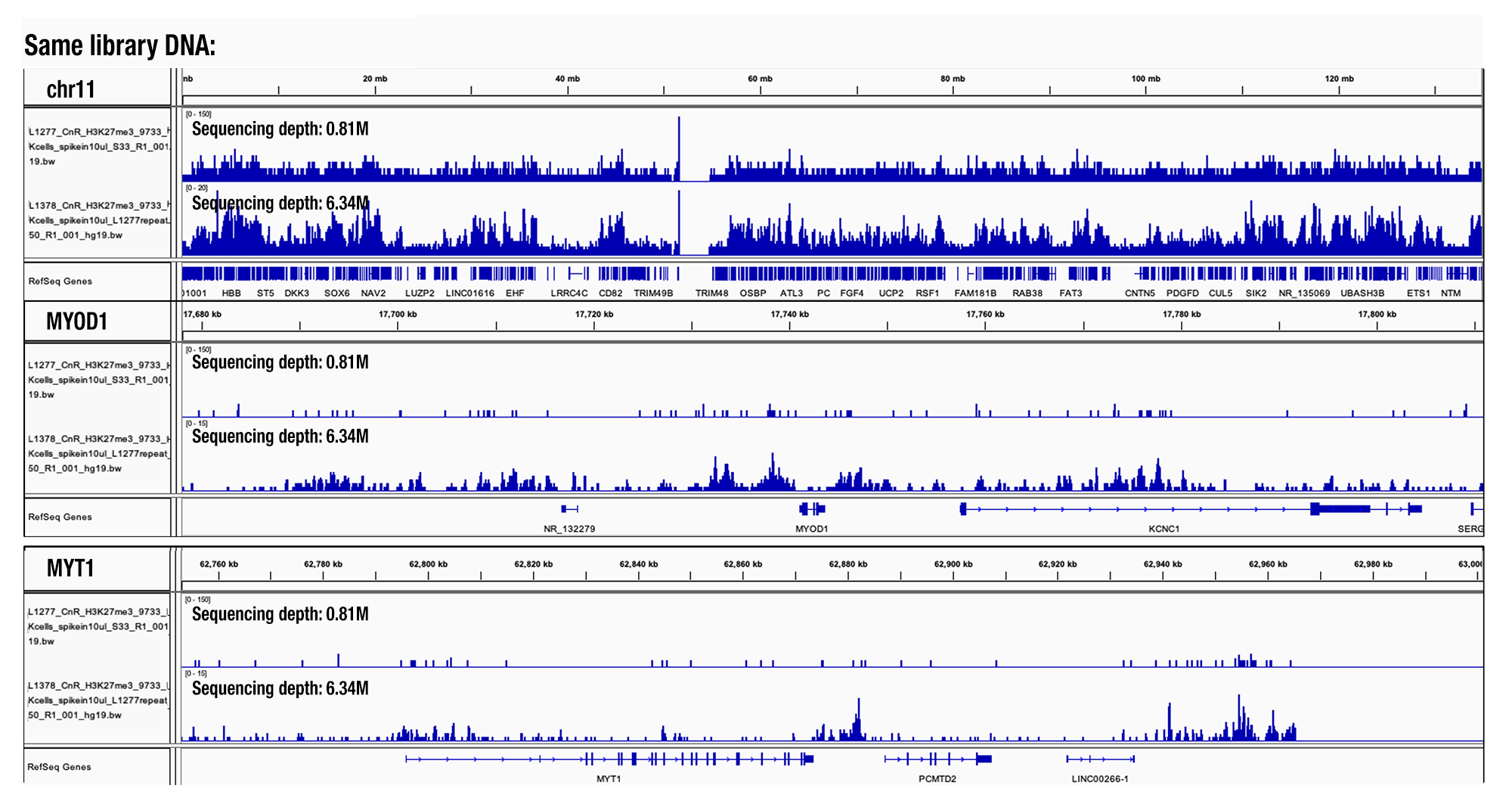
测序深度比较,目标 H3K27me3: 使用 CUT&RUN 检测试剂盒 # 86652,通过 HeLa 细胞和 Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb #9733 进行 CUT&RUN 分析。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。如所示,将相同的 DNA 库以不同的测序深度送入 NGS。该图显示了染色体 11(顶部轨道),MYOD1 基因(中间轨道)和 MYT1 基因(底部轨道)内的结合。
如有任何其他问题,请通过[受保护的电子邮件]直接联系我们的表观遗传学应用组。
Christopher Fry - 博士
表观遗传学产品开发总监
Fang Chen, PhD
表观遗传学应用小组组长
发布时间 2021 年 10 月