超声处理染色质免疫沉淀法实验步骤
产品专有: SimpleChIP® Plus Sonication Chromatin IP Kit #56383.
I. 组织交联和样品制备
收获组织时,去除样品中不需要的物质,如脂肪和坏死物质。组织可以立即处理和交联,或放在干冰上冷冻供后续处理。为获得最佳 ChIP 结果,每次免疫沉淀使用 25 mg 组织。应额外处理 5 mg 组织用于“分析染色质消化和浓度”,并用作输入染色质(第四部分)。不同组织类型的染色质产率不尽相同,每次免疫沉淀时,某些组织可能需要超过 25 mg。
一次染色质制备量规定为 100 至 150 mg 组织。对于某些组织类型,这种建议的组织量可能会导致低产率,但在超声处理过程中可确保高效的染色质碎裂。有关不同组织类型的预期染色质产率的更多信息,请参见附录 A。
在开始之前:
- 取出并加热 200X Protease Inhibitor Cocktail (PIC) #7012 和 Glycine Solution (10X) #7005。确保 PIC 完全解冻。
- 对于每份染色质制备物,配制 3 ml 磷酸盐缓冲盐水 (PBS) + 15 µl 200X PIC,并将它们放在冰上。
- 对于每份染色质制备物,配制 1 ml 1X ChIP Sonication Cell Lysis Buffer(0.5 ml 2X ChIP Sonication Cell Lysis Buffer #96529 + 0.5 ml 水)+ 5 µl 200X PIC,并将它们放在冰上。
- 对于每份染色质制备物,配制 28 µl 37% 甲醛,并保存在室温下。或者,可以使用 62.5 µl 16% 不含甲醇的甲醛。使用在制造商标示的有效期内的新鲜甲醛。
- 称重新鲜或冷冻的组织样品。对于每份染色质制备物,使用 100-150 mg 组织。
- 将组织样品放在皮氏培养皿中,并用干净的手术刀或剃须刀片切成 1-2 mm 的方块。将培养皿放在冰上。重要的是将组织置于低温下以防止蛋白质降解。
- 将切好的组织转移到 15 ml 锥形试管中,对于每份染色质制备物,加入 1 ml 冰冷的 PBS + PIC。
- 为了让蛋白与 DNA 交联,每 1 ml PBS + PIC中加入28 μl 37% 甲醛或 62.5 µl 16% 不含甲醇的甲醛,并在室温下放置至少 10 分钟。甲醛的最终浓度是 1%。对于组蛋白修饰 ChIP,固定 10 分钟即可;对于转录因子 ChIP,我们建议固定 10 至 30 分钟;而对于转录辅因子 ChIP,我们则建议固定 30 分钟(参见附件 B 中的图 7)。
- 每 1 ml PBS + PIC 加入 100 μl 10X Glycine #7005 来终止交联。混合后放在冰上孵育 5 分钟。
- 在 4°C 下以 1,200 x g 的离心力离心分离组织 5 分钟。
- 对于每份染色质制备物,去除上清液,并用 1 ml 冰冷的 PBS + PIC 洗涤。
- 在 4°C 下以 1,200 x g 的离心力离心分离 5 分钟。
- 重复一次步骤 7 和 8。
- 对于每份染色质制备物,去除上清液,并在 1 ml 1X ChIP Sonication Cell Lysis Buffer + PIC 中重悬组织。
- 使用切割移液管吸头将组织悬浮液转移到杜恩斯匀浆器中。
- 使用完全配套的研杵(类型 A)捣碎 20 次以分散组织块或直至观察到没有组织块。
- 将细胞悬浮液转移到 1.5 ml 试管中,随后立即进行胞核制备和染色质碎裂(第三部分)。
II. 细胞培养物交联和样品制备
为获得最佳 ChIP 结果,每次免疫沉淀使用大约 4 x 106 个细胞。对于 HCT 116 细胞,这相当于 15 cm 培养皿所含细胞(在 20 ml 生长培养基中的融合度为 90%)的 1/3。应额外处理 1 x 106 个细胞用于“分析染色质消化和浓度”,并用作输入染色质(第四部分)。
一次染色质制备量规定为 1 x 107 至 2 x 107 个细胞。对于某些细胞类型,这种建议的细胞量可能会导致低产率,但在超声处理过程中可确保高效的染色质碎裂。
在开始之前:
- 取出并加热 200X Protease Inhibitor Cocktail (PIC) #7012 和 Glycine Solution(10X) #7005。确保 PIC 完全解冻。
- 对于每个待处理的 15 cm 培养皿,配制 2 ml 磷酸盐缓冲液 (PBS) + 10 μl 200X PIC,并放在冰上。
- 对于每个待处理的 15 cm 培养皿,配制 40 ml PBS,并放在冰上。
- 对于每个待处理的 15 cm 细胞培养皿,配制 540 μl 37% 甲醛,并保存在室温下。或者,可以使用 1.25 ml 16% 不含甲醇的甲醛。使用在制造商标示的有效期内的新鲜甲醛。
- 对于每份染色质制备物,配制 1 ml 1X ChIP Sonication Cell Lysis Buffer(0.5 ml 2X ChIP Sonication Cell Lysis Buffer #96529 + 0.5 ml 水)+ 5 µl 200X PIC。
- 为让蛋白与 DNA 交联,每个含 20 ml 培养基的 15 cm 培养皿添加 540 μl 37% 甲醛或 1.25 ml 16% 不含甲醇的甲醛。短暂旋转混匀并置于室温下孵育 10 分钟。甲醛的最终浓度是 1%。添加甲醛可能导致培养基颜色发生变化。
- 每个含 20 ml 培养基的 15 cm 培养皿添加 2 ml 10X 甘氨酸,稍微涡旋混合,并在室温下孵育 5 分钟。添加甘氨酸可能导致培养基颜色发生变化。
- 对于悬浮细胞:
- 将细胞转移到 50 ml 锥形试管中,在 4℃ 下以 1,000 x g 的离心力离心分离 5 分钟,随后沉淀物用 20 ml 冰冷的 PBS 洗涤两次。去除 PBS 后继续进行步骤 3b。或者,细胞沉淀物可以放在干冰上冷冻,并保存在 -80°C 下供后续使用。
- 对于每份染色质制备物,在每 1 ml 1X ChIP Sonication Cell Lysis Buffer + PIC 中重悬最多 2 x 107 个细胞,并立即进行胞核制备和染色质碎裂(第三部分)。
- 对于黏附细胞:
- 去除培养基,细胞用 20 ml 冰冷的 1X PBS 洗涤两次,每次完全去除培养皿中的洗液。
- 每个 15 cm 培养皿添加 2 ml 冰冷的 PBS + PIC。将细胞刮入冷的缓冲液中。将所有培养皿的细胞混合放入一个 15 ml 锥形试管。
- 细胞在 4°C 下以 1,000 x g 的离心力离心分离 5 分钟。去除 PBS 后继续进行步骤 4d。或者,细胞沉淀物可以放在干冰上冷冻,并保存在 -80°C 下供后续使用。
- 对于每份染色质制备物,在每 1 ml 1X ChIP Sonication Cell Lysis Buffer + PIC 中重悬最多 2 x 107 个细胞,并立即进行胞核制备和染色质碎裂(第三部分)。
III. 胞核制备和染色质碎裂
一次染色质制备量规定为 100-150 mg 组织或 1 x 107-2 x 107 个组织培养细胞。可以同时进行多份染色质制备,只要相应增加缓冲液用量并对 1 ml 样品进行超声处理。超声处理所使用的细胞数量和样品量对于产生合适大小的染色质片段非常关键。
在开始之前:
- 取出并加热 200X Protease Inhibitor Cocktail (PIC) #7012。使用前确保它完全融化。
- 对于每份染色质制备物,配制 1 ml 1X ChIP Sonication Cell Lysis Buffer(0.5 ml 2X ChIP Sonication Cell Lysis Buffer #96529 + 0.5 ml 水)+ 5 µl 200X PIC。
- 对于每份染色质制备物,配制 1 ml ChIP Sonication Nuclear Lysis Buffer #28778 + 5 µl 200X PIC。
- 将在第一部分或第二部分配制的 1X ChIP Sonication Cell Lysis Buffer + PIC 中的细胞悬浮液放在冰上孵育 10 分钟。
- 在 4°C 下以 5,000 x g 的离心力让细胞沉淀 5 分钟。对于每份染色质制备物,去除上清液,然后在 1 ml 冰冷的 1X ChIP Sonication Cell Lysis Buffer + PIC 中再次重悬沉淀物。
- 细胞悬浮液放在冰上孵育 5 分钟。在 4°C 下以 5,000 x g 的离心力让细胞沉淀 5 分钟。请注意,超声处理之前,交联细胞可能并未完全裂解。
- 对于每份染色质制备物,在 1 ml 冰冷的 ChIP Sonication Nuclear Lysis Buffer #28778 + PIC 中重悬细胞,然后放在冰上孵育 10 分钟。将 1 ml 细胞悬浮液转移到大小合适的试管中进行超声处理。请注意,超声处理之前,交联细胞和胞核可能并未完全裂解。
- 通过超声处理碎片化染色质超声处理条件可能需要通过测试不同超声波仪功率设置和超声处理时长来凭经验确定。最佳超声处理条件可产生 60-90 % 小于 1kb 的染色质片段。较长的交联时间可能会使小于 1 kb 的片段比例降低至 30-60%,(见附件 B 中的图 7 和附件 C 中的图 8)。使用能产生所需染色质片段长度需要的最少超声处理循环次数,因为过度超声处理会因染色质苛刻处理而导致信号减弱或丢失。
- 对于每份声处理样品,我们建议每 1 ml ChIP Sonication Nuclear Lysis Buffer 使用 100-150 mg 组织或 1 x 107-2 x 107 个细胞。对更多和/或更浓的细胞进行超声处理会降低染色质碎裂的效率。
- 使用配有 1/8 英寸微探头的 Branson Digital Sonifier D250 探头超声波仪时,应用 8 分钟的 1 秒开/1 秒闭声处理周期(超声处理时间为 4 分钟)和 50% 振幅通常能产生良好的碎裂性能和染色质免疫沉淀效率。
- 请确保在超声处理时以及各处理步骤之间将含有染色质的试管置于冰水槽中,以便让染色质样品在超声处理期间保持冷却。切勿让探头接触到试管底部或试管壁。如果染色质样品在超声处理期间起泡,则停止超声处理并调整试管位置。
- 在 4°C 下用微量离心机以 21,000 x g 的离心力离心分离 10 分钟来使裂解物变澄清。
- 将上清液转移至新试管。所得到的就是交联染色质制备物,可以立即用于进行免疫沉淀或保存在 -80°C 下供后续使用。取出 50 μl 染色质制备物进行染色质消化和浓度分析(第四部分)。
IV. 分析染色质碎裂和浓度(建议步骤)
- 向 50 μl 染色质样品(在第 三 部分的步骤 7 中制备)中添加 100 μl 无核酸酶水、6 μl 5 M NaCl #7010 和 2 µl RNAse A#7013。涡旋混合,并在 37℃ 下孵育样品 30 分钟。
- 向每份经 RNase A 消化的样品添加 2 μl Proteinase K #10012。涡旋混合并在 65℃ 下孵育样品 2 小时。
- 按第七部分中所述的步骤,使用 DNA 纯化离心柱从样品中纯化 DNA。
- DNA 纯化后,取 10 μl 样品并通过在 1% 琼脂糖凝胶上进行电泳来确定 DNA 片段大小。DNA 拖尾预计为 200 bp 到几 kb(见附件 C 中的图 7)。大约 60-90% 的所有 DNA 片段应小于 1 kb。请参见附件 B 了解超声处理条件的优化。
- 使用分光光度计测量 DNA 浓度。在理想情况下,DNA 浓度应介于 50 和 200 µg/ml 之间。
V. 染色质免疫沉淀法
为获得最佳 ChIP 结果,每次免疫沉淀使用大约 5-10 μg 经超声处理的交联染色质(第 四 部分中确定的量)。这应大致相当于用 25 mg 离散组织或 4x106 个组织培养细胞制备的一份 100 µl 免疫沉淀制备物。在添加抗体之前,通常将 100μl 消化的染色质稀释到 400μl 1X ChIP 缓冲液中。但如果每次免疫沉淀需要多于 100 µl 的染色质,则必须将交联染色质制备物按 1:4 的稀释比例在 1X ChIP 缓冲液中稀释。在这种情况下,无需添加 蛋白 G 磁珠,尽管延长用微珠孵育的时间比较有用。
在开始之前:
- 取出并加热 200X Protease Inhibitor Cocktail (PIC) #7012。确保 PIC 完全解冻。
- 取出并加热 10X ChIP Buffer #7008,确保 SDS 完全溶解。
- 融化碎裂的染色质制备物(在第 三 部分的步骤 7 中制备)并放在冰上。
- 制备低盐洗液:每次免疫沉淀用 3 ml 1X ChIP Buffer(300 µl 10X ChIP Buffer #7008 + 2.7 ml 水)。置于冰上。
- 制备高盐洗液:每次免疫沉淀用 1 ml 1X ChIP Buffer(100 µl 10X ChIP Buffer #7008 + 900 µl 水)+ 70 µl 5M NaCl #7010。置于冰上。
注:对于 Cell Signaling Technology 的多数抗体,每份免疫沉淀样品用 1- 2 µg 时效果最佳。如有多份不同浓度的样品,最好让阴性对照 Normal Rabbit IgG #2729 与最高抗体浓度相匹配。
- 在一根试管中,配制足够的 1X ChIP Buffer + PIC,用于将经超声处理的染色质稀释至能进行所需次数的免疫沉淀。对于每次免疫沉淀,染色质(5-10 µg DNA)用 1X ChIP Buffer + PIC 稀释至总量为 500 µl。要进行高效的免疫沉淀,必须以 1:4 或更高的稀释因数在 1 x ChIP buffer 中稀释染色质。将混合物置于冰上。确定免疫沉淀的次数时,记得计入阳性对照样品 Histone H3 (D2B12) XP® Rabbit mAb#4620 和阴性对照样品 Normal Rabbit IgG antibody #2729。
- 取出 10 μl 稀释的染色质样品并将其转移至微量离心管。这将作为 2% 样品输入对照,该样品在后续使用之前可以置于 -20℃ 下贮存(第六部分的步骤 1 )。
- 对于每次免疫沉淀,将 500 μl 稀释的染色质分装到 1.5 ml 微量离心管中,并加入免疫沉淀抗体。每次免疫沉淀所需的抗体用量有所不同,应按照制造商的建议使用。对于阳性对照 Histone H3 (D2B12) XP® Rabbit mAb#4620,向免疫沉淀样品加入 10 μl。对于阴性对照 Normal Rabbit IgG #2729,向免疫沉淀样品加入 1 µl (1 µg) 至 2 µl (2 µg)。免疫沉淀样品在 4℃ 下振荡孵育 4 小时至过夜。
- 轻轻涡旋来重悬 ChIP-Grade Protein G Magnetic Beads #9006。每次免疫沉淀反应立即添加 30 µl Protein G Magnetic Beads,并在 4°C 下振荡孵育 2 小时。或者,微珠可以添加到步骤 3 中的过夜抗体孵育液中,但这可能会增强背景信号。
- 在每次免疫沉淀中,将试管放在磁分离架上让 Protein G Magnetic Beads 沉淀。等待 1 至 2 分钟直到溶液澄清,然后小心地移除上清液。
- Protein G Magnetic Beads 通过添加 1 ml 低盐洗液到微珠中进行洗涤,并在 4°C 下振荡孵育 5 分钟。再重复步骤 6 和 7 两次,以确保总共进行 3 次低盐洗涤。
- 向微珠中添加 1 ml 高盐洗液并且在 4℃ 下转动孵育 5 分钟。
- 在每次免疫沉淀中,将试管放在磁分离架上让 Protein G Magnetic Beads 沉淀。等待 1 至 2 分钟直到溶液澄清,然后小心地移除上清液。立即进入第六部分。
VI. 将染色质从抗体/Protein G Magnetic Beads 上洗脱下来并解交联
在开始之前:
- 取 2X ChIP Elution Buffer #7009 放在 37°C 水浴槽中加热,并确保 SDS 溶解。
- 将水浴或热混合仪设为 65℃。
- 配制每次免疫沉淀所需的 150 µl 1X ChIP Elution Buffer(75 µl 2X ChIP Elution Buffer #7009 + 75 µl 水)和 2% 输入样品。
- 添加 150 µl 1X ChIP Elution Buffer 到 2% 输入样品试管中,并放在室温下直至进行步骤 7。
- 向每份 IP 样品中添加 150 μl 1X ChIP 洗脱缓冲液。
- 在 65°C 下,通过轻轻涡旋 (1,200 rpm) 将染色质从抗体/Protein G Magnetic Beads 上洗脱下来,持续 30 分钟。这个步骤最好使用恒温混匀仪,但在 65°C 水浴槽中频繁混合也可。或者,可以在室温下转动洗脱,但是可能不会完全洗脱。
- 样品以 10,000 x g 的离心力短暂离心分离 10 秒钟,以便去除微量离心管盖上的蒸发样品。
- 将试管放在磁分离架上让 Protein G Magnetic Beads 沉淀,并等待 1-2 分钟让溶液变澄清。
- 小心地将洗脱下来的染色质上清液转移至新试管。
- 要解交联,向所有试管(包括步骤 1 中的 2% 输入样品)加入 6 µl 5M NaCl #7010 和 2 µl Proteinase K #10012,并在 65°C 下孵育 2 小时。这次孵育可以延长过夜。
- 立即进入第七部分。或者,样品可在 -20℃ 下贮存。但为了避免形成沉淀物,请确保在添加 DNA Binding Buffer #10007 之前,将样品加热到室温(第 七 部分,步骤 1)。
VII. 使用离心柱纯化 DNA:
在开始之前:
- 使用前添加 24 ml 乙醇 (96-100%) 到 DNA Wash Buffer #10008 中。此操作仅需在第一次DNA纯化前进行。
- 为第 六 部分的每份 DNA 样品准备一根 DNA 纯化离心柱和收集管 #10010。
- 添加 750 µl DNA Binding Buffer #10007 到每份 DNA 样品中,涡旋片刻。
- 对于每 1 体积样品,应当使用 5 倍体积的 DNA 结合缓冲液。
- 对于步骤 1 中的每份 DNA 样品,转移450 μl 到收集管中的 DNA 离心柱上。
- 于微量离心机中以 14,000 转/分钟离心 30 秒。
- 从收集管取出离心柱并丢弃液体。离心柱再放入收集管中。
- 对于步骤 1 中的每份 DNA 样品,转移剩余的 450 μl 到收集管中的离心柱上。重复步骤 3 和 4。
- 添加 750 µl DNA Wash Buffer #10008 到收集管中的离心柱上。
- 于微量离心机中以 14,000 转/分钟离心 30 秒。
- 从收集管取出离心柱并丢弃液体。离心柱再放入收集管中。
- 用微量离心机以 14,000 rpm 的转速离心分离 30 秒钟。
- 丢弃收集管和液体。保留离心柱。
- 添加 50 μl DNA Elution Buffer #10009 到每根离心柱上,并放在 1.5 ml 干净的微量离心管中。
- 于微量离心机中以 14,000 转/分钟离心 30 秒,以洗脱 DNA。
- 取出并丢弃 DNA 离心柱。洗脱物即纯化的 DNA。样品可在 -20℃ 下贮存。
VIII. 通过 PCR 定量 DNA:
建议:
- 使用过滤式吸液头来尽量减少污染风险。
- 该试剂盒中的对照引物对人或小鼠 RPL30 基因(#7014 或 #7015)具有特异性,可用于标准 PCR 或定量实时 PCR。如果用户对其他物种进行 ChIP,建议用户设计针对 DNA 的相应引物,并确定最佳 PCR 条件。
- 建议使用热启动 Taq 聚合酶以最大限度降低非特异性 PCR 产物的风险。
- PCR 引物选择至关重要。应当严格遵循以下标准设计引物:
| 引物长度: |
24 个核苷酸 |
| 最佳 Tm: |
60℃ |
| 最佳 GC: |
50% |
| 扩增子尺寸: |
150 至 200 bp(标准 PCR) |
| |
80 至 160 bp(实时荧光定量 PCR) |
标准 PCR 方法:
- 标记适当数量、兼容待用 PCR 仪型号的 PCR 管或 PCR 平板。这些样品应当包括 2% 样品输入对照、阳性对照组蛋白 H3 样品、阴性对照 Normal Rabbit IgG 样品,和未加入 DNA 的试管以排除 DNA 污染。
- 向每支管中添加 2 μl 相应的 DNA 样品。
- 按下文所述的步骤制备主反应混合物,配置时多配置两个样本的试剂以弥补分管时的体积损耗。向每支反应管中添加 18 μl 主混合物。
| 试剂 |
1 次 PCR 反应所需体积 (18 μl) |
| 无核酸酶的 H2O |
12.5 μl |
| 10X PCR 缓冲液 |
2.0 μl |
| 4mM dNTP 混合物 |
1.0 μl |
| 5 μM RPL30 引物 |
2.0 μl |
| Taq DNA 聚合酶 |
0.5 μl |
- 启动以下 PCR 反应程序:
- 初始变性:在 95°C 下进行 5 分钟
- 变性:在 95°C 下进行 30 秒钟
- 退火:在 62°C 下进行 30 秒钟
- 延伸:在 72°C 下进行 30 秒钟
- 重复步骤 b-d,共循环 34 次。
- 最终延伸:在 72°C 下进行5 分钟
- 从每支反应管中取出 10 μl PCR 产物,使其与 100 bp DNA 标准品同时进行 2% 琼脂糖凝胶或 10% 聚丙烯酰胺凝胶电泳,以进行分析。人 RPL30 #7014 和小鼠 RPL30 #7015 的 PCR 产物的预期大小分别是 161 bp 和 159 bp。
实时荧光定量 PCR 方法:
- 标记适当数量、兼容待用 PCR 仪型号的 PCR 管或 PCR 平板。PCR 反应应当包括阳性对照组蛋白 H3 样品、阴性对照 Normal Rabbit IgG 样品、监控 DNA 污染的不含 DNA 模板的试管和 2% 输入对照组染色质 DNA 的连续稀释物(未稀释、1:5、1:25、1:125),以生成标准曲线并测定扩增效率。
- 向 PCR 平板上的每只管或每个孔中添加 2 μl 相应的 DNA 样品。
- 按下文所述的步骤制备主反应混合物。每次 PCR 反应设置 2-3 次复制。添加足够试剂来补充容积损失。向每支 PCR 反应管或每个孔中添加 18 μl 反应混合物。1 次 PCR 反应的试剂量 (18 μl)
| 无核酸酶的 H2O |
6 μl |
| 5 μM RPL30 引物 |
2 μl |
| SimpleChIP® Universal qPCR Master Mix #88989 |
10 μl |
- 启动以下 PCR 反应程序:
| a. |
初始变性 |
95℃ 3 分钟 |
| b. |
变性 |
95℃ 15 秒 |
| c. |
复性和延伸: |
60℃ 60 秒 |
| d. |
重复步骤 b 和 c,共循环 40 次。 |
|
- 使用实时 PCR 仪的自带软件,分析定量 PCR 结果。或者,可以使用input样品百分数方法和以下所示的公式,手动计算 IP 效率。采用这种方法,从每次免疫沉淀获得的信号表述为占总input样品染色质的百分比。
输入百分比 = 2% x 2(C[T] 2% 输入样品 - C[T] 免疫沉淀样品)
C[T] = CT = PCR 反应的平均循环阈值
IX. NG-Sequencing文库构建
用这种试剂盒制备的免疫富集的 DNA 样品可用于 ChIP-seq 。要构建下游 NG 测序用 DNA 文库,请使用与您的下游测序平台相容的 DNA 文库制备实验步骤或试剂盒。对于 Illumina® 平台上测序,我们建议使用 DNA Library Prep Kit for Illumina® (ChIP-seq, CUT&RUN) #56795 及其相关索引引物 Multiplex Oligos for Illumina® (Single Index Primers) (ChIP-seq, CUT&RUN) #29580 或 Multiplex Oligos for Illumina® (Dual Index Primers) (ChIP-seq, CUT&RUN) #47538。
建议:
- 对于转录因子或辅因子 ChIP-seq,使用至少 5 ng ChIP 富集的 DNA 和 10 个 PCR 循环扩增衔接子连接的 DNA。
- 对于总组蛋白和组蛋白修饰或样品输入对照,开始时使用至少 50ng ChIP 富集的 DNA 和 6 个 PCR 循环扩增衔接子连接的 DNA。
- 要对全部靶标类型的 ChIP-富集的 DNA 构建文库,需对衔接子连接的 DNA 进行纯化,但无需进行大小选择。
- 在构建 DNA 文库之后,使用 Agilent High Sensitivity DNA Kit (Agilent Technologies,货号:G2938-90322)或通过与 50-100 ng DNA 在 2% 琼脂糖 TAE 凝胶上进行琼脂糖凝胶电泳,来检验 DNA 文库中衔接子二聚体(约 140 bp)是否存在。如果有接头蛋白二聚体,则重复清除 PCR 扩增物质。
- 也可通过对已知阳性和阴性靶基因座使用 qPCR 和引物组来确认文库的质量。与阴性引物相比,阳性引物对仍然应当产生相同的高信号,如原始 qPCR 分析 ChIP 富集的 DNA 时所见。
- 完成最终的纯化和质量检验后,制备浓度为 2-10 nM 的最终纯化文库样品以用于高通量测序。
附录 A:预期的染色质产率
从组织样品收获交联的染色质时,组织类型之间的染色质产率可能显著不同。右表提供用 100 mg 组织与 2 x 107 个 HCT 116 细胞制备的染色质的预期产量范围,以及实验步骤第 四 部分确定的预期 DNA 浓度。为了获得最佳 ChIP 结果,我们建议使用 DNA Library Prep Kit for Illumina® (ChIP-seq, CUT&RUN) #56795 及其相关索引引物 Multiplex Oligos for Illumina® (Single Index Primers) (ChIP-seq, CUT&RUN) #29580 或 Multiplex Oligos for Illumina® (Dual Index Primers) (ChIP-seq, CUT&RUN) #47538。
| 组织/细胞 |
染色质总产率 |
预期 DNA 浓度 |
| 肝 |
每 100 mg 组织使用 50 µg |
150 µg/ml |
| 脑 |
每 100 mg 组织使用 25 µg |
50 µg/ml |
| 心脏 |
每 100 mg 组织使用 105 µg |
20 µg/ml |
| HCT 116 |
100-150 μg/2 x 107 个细胞 |
100-150 μg/ml |
附录 B:甲醛固定的优化
转录因子和辅因子结合染色质 DNA 没有结合组蛋白紧密。因此,它们在超声处理期间容易脱离染色质。在 ChIP 实验中,延长固定时间能捕获更多转录因子和辅因子,尤其是使用组织样品时。如图 7 所示,固定时间从 10 分钟延长至 30 分钟可能会减小染色质片段(左小图),但 ChIP-qPCR 表明,这会显著增加心脏组织中辅因子 RING1B 和 SUZ12 的富集(中间小图和右小图)。
通常,对于同时使用细胞和组织样品的组蛋白修饰 ChIP,10 分钟固定即可,而转录因子和辅因子可能需要长达 30 分钟的额外固定时间,尤其是使用组织样品时。
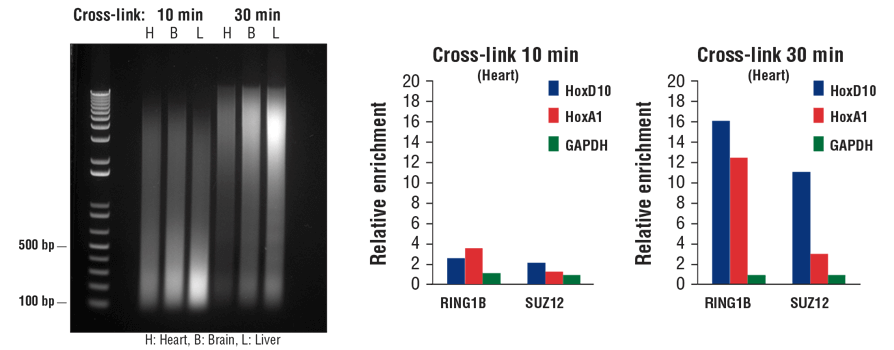
图 7. 使小鼠心脏 (H)、脑 (B) 和肝 (L) 细胞交联 10 分钟或 30 分钟,如图所示(左小图)。染色质制备后进行超声处理,DNA 进行纯化并通过对 1% 琼脂糖凝胶进行电泳来分离 20 µl。在 ChIP-qPCR 检测(中间小图和右小图)中,使用 10 µl RING1B (D22F2) XP® Rabbit mAb #5694 或 5 µl SUZ12 (D39F6) XP® Rabbit mAb #3737 进行染色质免疫沉淀。使用 SimpleChIP® Mouse HoxD10 Exon 1 Primers #7429、SimpleChIP® Mouse HoxA1 Promoter Primers #7341 和 SimpleChIP® Mouse GAPDH Intron 2 Primers #8986,通过实时 PCR 对富集的 DNA 进行定量分析。每份样品中免疫沉淀 DNA 的数量表示为向阴性 GAPDH 基因座发出的标准化信号(等于 1)。
附录 C:染色质碎裂的优化
交联染色质 DNA 碎裂的最佳条件高度依赖于使用的细胞数量、样品体积、超声处理时长和超声波仪功率设置。对于每份声处理样品,我们建议每 1 ml ChIP Sonication Nuclear Lysis Buffer 使用 100-150 mg 组织或 1 x 107 - 2 x 107 个细胞。 以下是确定某种特定组织或细胞类型的最佳超声处理条件的实验步骤。
- 按照第 一、二 和 三 部分的说明,用 100-150 mg 组织或 1 x 107-2 x 107 个细胞制备交联胞核。在第三部分的步骤 4 之后停止,并如下所述继续。
- 通过超声处理碎片化染色质对于指定超声波仪,可以通过在规定功率设置(参见第 三 部分的步骤 5,以了解使用 Branson Digital Sonifier 250 探头超声波仪的最佳功率设置)下改变超声处理循环次数或时长来确定最佳超声处理条件。要确定最佳超声处理条件,设计一项超声处理时程实验,并在某个超声处理循环或时长后取出 50 µl 染色质样品。例如,每 1-2 分钟超声处理后取出染色质样品。
- 在 4°C 下用微量离心机以 21,000 x g 的离心力离心分离 10 分钟,以让染色质样品变澄清。
- 将上清液转移到新微量离心管中,并加入 100 µl 无核酸酶水、6 µl 5 M NaCl #7010 和 2 µl RNAse A #7013。涡旋混合,并在 37℃ 下孵育样品 30 分钟。
- 向每份经 RNase A 消化的样品添加 2 μl Proteinase K #10012。涡旋混合并在 65℃ 下孵育样品 2 小时。
- 从每份样品中取 20 µl,并确定 DNA 片段大小。
- 选择能产生最佳 DNA 片段大小的超声处理条件,并用于第 三 部分步骤 5 中的染色质制备。如果未达到最佳超声处理条件,则增加或减少超声波仪的功率设置,并重复超声处理时程。
注:最佳超声处理条件根据样品类型和固定时间而不同。使用产生所需长度染色质片段需要的最少超声处理循环次数。超过 80% 短于 500 bp 的总 DNA 片段表明,过分超声处理会导致染色质过度受损,并会降低免疫沉淀效率(见图 8,右小图)
- 对固定 10 分钟的细胞进行超声处理时,最佳超声处理条件会产生 DNA 拖尾,其中含有约 90% 短于 1 kb 的 DNA 总片段(见图 8,左小图)。延长固定时间至 30 分钟会限制碎裂过程,从而产生 DNA 拖尾,其中含有约 60% 短于 1 kb 的 DNA 总片段。
- 对固定 10 分钟的组织进行超声处理时,最佳超声处理条件会产生 DNA 拖尾,其中含有约 60% 短于 1 kb 的 DNA 总片段。延长固定时间至 30 分钟会限制碎裂过程,从而产生 DNA 拖尾,其中含有约 30% 短于 1 kb 的 DNA 总片段(见图 7,左小图)。
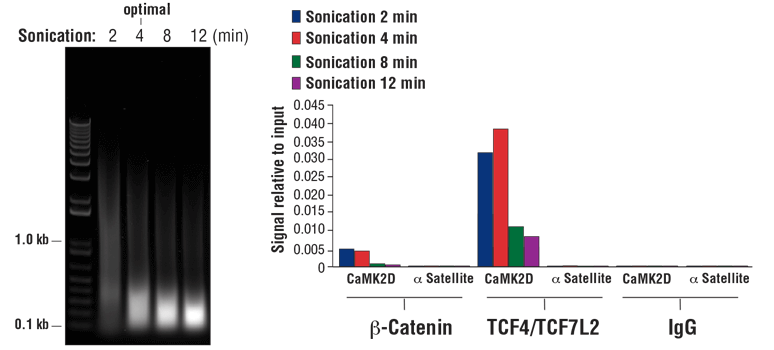
图 8. 对交联 10 分钟并经超声处理规定时长的 2 x 107 个 HCT 116 细胞进行染色质免疫沉淀(左小图)。DNA 按照 SimpleChIP® Plus Sonication Chromatin IP Kit #56383 的第 四 部分的说明进行纯化,并在 1% 琼脂糖凝胶上进行电泳来分离 20 µl 纯化 DNA。如左小图所示,增加超声处理循环次数会减小染色质片段的大小。使用 SimpleChIP® Plus Sonication Chromatin IP Kit #56383 对 5 µl Non-phospho (Active) Β-Catenin (Ser33/37/Thr41) (D13A1) Rabbit mAb #8814、10 µl TCF4/TCF7L2 (C48H11) Rabbit mAb #2569 或 2 µl Normal Rabbit IgG #2729 进行染色质免疫沉淀。使用 SimpleChIP® Human CaMK2D Intron 3 Primers #5111 和 SimpleChIP® Human α Satellite Repeat Primers #4486,通过实时 PCR 对富集的 DNA 进行定量分析。每份样品中免疫沉淀 DNA 的数量表示为与输入染色质的总量(等于 1;右小图)相对应的信号。如图所示,使用配有 1/8 英寸微探头的 Branson Digital Sonifier D250 探头超声波仪时,4 分钟的染色质声处理最佳。过度超声处理会明显有损辅因子 β-catenin 和包含染色质的转录因子 TCF4/TCF7L2 的富集。
附录 D:疑难排解指南
将交联时间缩短到 10-30 分钟的范围。减少每次超声处理的细胞/组织数量。进行超声处理时程。
| 问题 |
可能的原因 |
建议 |
| 1. 碎裂染色质的浓度过低。 |
细胞/细胞核裂解不完全。
染色质制备没有使用足够细胞。
|
如果染色质制备物的 DNA 浓度接近于 50 μg/ml,则向每次 IP 中添加额外的染色质以确保每次 IP 至少产生 5 μg,然后继续实验。
交联前,对单独平板上的细胞进行计数,以确定准确的细胞数量。
|
| 2. 染色质碎裂不足,且片段过大(超过 50% 的片段大于 1.5 kb)。 |
细胞可能已经过度交联。
处理了过多细胞/组织。
|
将交联时间缩短到 10-30 分钟的范围。减少每次超声处理的细胞/组织数量。进行超声处理时程。
|
| 3. 染色质过度碎裂(超过 90% 的片段小于 300 bp)。 |
超声处理条件太苛刻。
|
进行一次超声处理时程来找到进行相应超声处理的最小输出/时长。
|
| 4. 样品输入对照组 PCR 反应中无产物或产物很少。 |
添加至 PCR 反应的 DNA 不足或条件不是最佳。
PCR 扩增区域可能跨越无核小体的区域。
加入到免疫沉淀的染色质不足,或染色质被过度超声处理。
|
向 PCR 反应中添加更多 DNA 或增加扩增循环次数。
使用从交联并经超声处理的染色质中获得的纯化 DNA 来优化针对实验用引物组的 PCR 条件。为获得最佳 ChIP 结果,每次 IP 添加 5-10 μg 染色质。参见上述问题 1 和 3 的建议。
|
| 5. 阳性对照组蛋白 H3-IP RPL30 PCR 反应中无产物。 |
添加至 IP 反应的染色质或抗体不足,或者 IP 孵育时间过短。
蛋白 G 微珠中染色质洗脱不完全。
|
确保每次免疫沉淀反应添加 5-10 µg 染色质和 10 µl 抗体,并用抗体孵育过夜,添加蛋白 G 微珠之后再孵育 2 小时。
在 65℃ 下将染色质从蛋白 G 微珠洗脱为最佳,同时频繁混合以保持微珠悬浮在溶液中。
|
| 6. 阴性对照 Rabbit IgG-IP 和阳性对照 Histone H3-IP PCR 反应中产物的数量相等。 |
添加至 IP 反应的染色质过多或不足。或者,添加至 IP 反应的抗体过多。
添加至 PCR 反应的 DNA 过多或扩增循环次数过多。
|
向每次 IP 反应中添加不超过 15 μg 的染色质和 10 μl Histone H3 Antibody。每次 IP 将 Normal Rabbit IgG 缩减至1 µl/IP。
向 PCR 反应中添加更少的 DNA 或减少 PCR 循环次数。在 PCR 的线性扩增阶段范围内分析 PCR 产物极为重要。否则,起始 DNA 数量的差异无法准确测量。
|
| 7. 实验抗体 IP PCR 反应中无产物。 |
添加至 PCR 反应的 DNA 不足。
添加至 IP 反应的抗体不足。
抗体不适用于 IP。
|
向 PCR 反应中添加更多 DNA 或增加扩增循环次数。
通常,免疫沉淀反应需添加 1-5 µg 抗体;但准确的用量在很大程度上取决于具体抗体。
增加添加至 IP 反应的抗体量。寻找其他替代抗体。
|
发布时间 2017 年 3 月
修订时间 2022 年 4 月

